0 引言
水稻育种对维护全球粮食安全有重要意义,近几十年中国在水稻育种领域有重要贡献[1-2]。随着育种技术的发展,不断更新的育种知识越发需要传播。问答系统是目前传播知识的应用方向之一,可以帮助科技工作者传播新兴技术,降低知识获取难度,为知识传播提供便利。近年来已经存在一些水稻知识问答方法。文献[3]提出了一种基于词向量模型(Word embeddings, Word2vec)和长短期记忆网络(Long short-term memory, LSTM)的句子相似度模型,提高了水稻句子相似度计算的精确率。文献[4]提出了一种基于残差LSTM和序列到序列模型(Sequence to sequence, Seq2Seq)的水稻问答模型,提高了模型输出与实际问句的相似度。然而,水稻育种方面仍存在数据存储分散、信息管理技术落后以及知识粒度大等问题,这使得问答系统难以输出语言组织较好的答案。现有置信度高的水稻在线数据库有“Ricedata”、“Hybridrice”和“Cgris”。这些数据库可以提供品种查询功能,但存在检索不完备和知识粒度大等问题。为了提高水稻育种数据管理水平,文献[5]结合育种数据开发了水稻信息管理系统,提高了水稻数据组织性。文献[6]提出了一种基于本体的水稻育种知识库的构建方法,提高了知识库分析用户问题的能力。文献[7]基于BERT(Bidirectional encoder representation from transformers)模型提出了一种针对知识图谱中水稻表型组学实体的关系分类方法,显著提高了关系抽取的精度。然而由于传统数据库存储方式的限制,当前水稻育种知识应用水平仍有待提高,水稻育种领域需要引入更高效的数据管理方法。
知识图谱是近年来新兴的一种知识表示技术。该技术已被证明在垂直领域中对知识管理和数据分析具有革新意义[8]。相比于传统的关系型数据库,知识图谱[9]主要以图数据库为载体,在水稻育种问答系统中可以更高效地管理大规模的领域数据[10-11]。知识图谱以三元组形式(实体-关系-实体)进行知识表示,且具有可修改和遍历的边[12-13]。这使得知识图谱可以统一水稻育种数据和映射不同实体之间的联系。高质量通用知识图谱已经有很多,百科类知识图谱有“Wikidata”、“CN-DBpedia”以及“BabelNet”等。然而垂直领域知识图谱由于涉及行业数据隐私,获取深度数据难,导致构建困难,并且公开较少[14]。目前知识图谱在水稻领域有一些应用案例[15-16]。然而,当前水稻育种领域信息检索和数据管理水平仍低下,构建水稻育种知识图谱成为进一步提高水稻育种知识管理和应用的一大需求。
为了进一步提高水稻育种知识管理水平,本文结合水稻育种数据和本体模型构建水稻育种知识图谱。结合现有水稻数据库文本结构以及专家知识,对水稻育种文本进一步划分,增加小类命名实体类别,在保证文本信息表述的前提下降低知识粒度。然而,由于缺乏可用的水稻育种标注数据,需要对水稻育种问句进行标注。随着命名实体类别的增加,高昂的标注成本造成难以获得大规模且高质量的标注数据。而对标注文本进行数据增强(Data augmentation, DA)恰恰能在降低标注成本的同时,获取到泛化性较好的标注数据。因此,DA技术的引入成为了提高水稻育种问句命名实体识别模型精确率的关键技术之一。
DA是提高训练模型效果,防止过拟合的一种技术,用于数据集小、样本相似度高、模型难以充分训练的情况。目前,由于在大多数语言和领域中用于命名实体识别(Named entity recognition, NER)的手动标记训练数据量是有限的,DA技术在自然语言处理(Natural language processing, NLP)领域已经有一些研究成果[17-21]。简单数据增强(Easy data augmentation, EDA)方法和实例替换方法由于可解释性强,实现成本低,且增强效果能媲美基于深度学习的生成语言模型,二者均适用于水稻育种问句的句式结构和命名实体分布特点。
BERT、双向长短期记忆模型(Bi-directional long short-term memory, BISLTM)以及条件随机场(Conditional random field, CRF)在农业领域文本的NER任务中已有应用。文献[22]提出利用CRF模型来识别农技问答数据中农作物、病虫害以及农药等命名实体,有效提高了识别的准确度。文献[23]提出了利用BERT-CRF模型来识别生鲜蛋供应链文本中命名实体,提高了模型在菜谱数据集的识别精确率。文献[24]提出利用融合多元特征信息的BERT-BILSTM-CRF模型来识别农作物病虫害文本,提高了模型捕捉上下文信息的能力。BERT层为预训练语言模型,可以学习到水稻问句中短语表示和语义特征[25]。BILSTM层为双向编码,可以学习到水稻育种连续问句的上下文联系[26]。CRF层作为条件随机场,可以学习到输出标注序列的隐藏约束条件[27]。3层的巧妙结合可以使得模型对水稻育种文本的NER效果有更高的性能[28]。为增强现有的标注数据,本文将结合EDA和实例替换的方法,在BERT-BILSTM-CRF模型上引入改进的数据增强层DA,在实例替换时加入水稻育种词汇实体,以增强不同类别的命名实体。
1 工作框架
为提高水稻育种问答系统的数据管理水平,根据水稻育种数据的文本分类和水稻育种问句的实体类型,结合水稻生长习性对水稻育种领域进行知识建模,构建了水稻育种本体模型。在构建本体模型时,将半结构化的水稻育种文本进一步划分,降低了知识粒度,根据本体模型将水稻育种数据通过图数据库进行结构化存储,构建成为水稻育种知识图谱。
将水稻育种知识图谱中的实体和实体属性映射到水稻育种问句的命名实体,并参照图谱中的实体和实体属性定义问句中的命名实体类别,在大类实体中进一步划分出小类实体,从而增加实体类别,以提高后续文本数据增强的效果。
针对当前缺乏水稻育种标注文本、人工标注成本高等问题,提出结合水稻育种问句命名实体类别在NER模型BERT-BILSTM-CRF中引入数据增强层DA。实验结合EDA和实例替换方法构建DA层,并融入水稻育种词汇信息,对水稻育种问句进行命名实体增强,之后通过增强的标注数据,来提高水稻育种问句NER模型的性能,具体如图1所示。
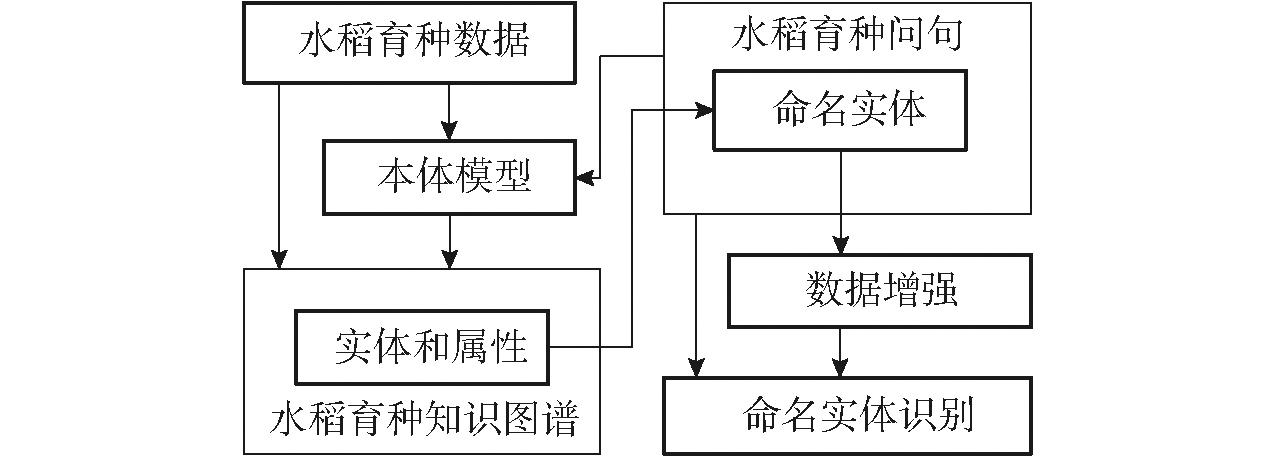
图1 研究框架
Fig.1 Research framework
2 数据获取与处理
2.1 数据获取
构建水稻育种知识图谱需要置信度高且覆盖性广的水稻育种数据。因而在选取数据来源时,相比于从文献或者书籍获取的方式,以存储不同水稻品种信息的水稻育种数据库为基准数据更具可行性。本文构建水稻育种知识图谱的数据来源于中国种业大数据平台,所存储的育种数据为半结构化文本。之后本文通过文本分类和实体类别划分将水稻育种数据转换为结构化的三元组,并采用主流的Neo4j图数据库(https:∥neo4j.com/)进行存储和表示,从而构成水稻育种知识图谱。所进行命名实体识别的水稻育种问句的原始文本数据来自于中国农技信息推广服务平台的问答社区(https:∥njtg.nercita.org.cn/user/index.shtml/),并且采用基于Scrapy框架的爬虫技术批量获取问句数据。
2.2 数据标注
为了节省水稻育种数据标注的人力成本和提高标注效率,采用精灵标注工具(http:∥www.colabeler.com/)对水稻育种问句逐句进行标注。选取的标注方式为主流且规则简单的BIO(Beginning, Inside, Outside)方式。其中在BIO标注规则里,“B-Label”代表“Label”类实体的首个字符,“I-Label”代表“Label”类实体的中间或结尾字符,“O”代表非实体的无关字符。标签“Label”即为人工标注时自行定义,为了便于标签的区分和理解,在标注水稻育种问句中的命名实体时,采用语义与实体类型相近或匹配的字符串作为标注的标签。例如“B-Characteristics”和“I-Characteristics”分别代表水稻植株特点实体第1个位置的字符和其他位置的字符。
3 知识图谱构建
构建知识图谱目前有两大类方法,自底向上和自顶向下。自底向上是从大量的数据文本中提取实体,然后把相似的实体归为一类。根据一些实体之间的联系抽象出关系和概念,将置信度高的实体和关系导入数据库,构成知识图谱。自顶向下是先根据需要存储的知识对象来定义概念和关系,然后定义知识图谱的本体模型。根据本体模型将知识进行划分,储存到数据库形成知识图谱。由于可获取到置信度高的半结构化的水稻育种数据,本文采用自顶向下的方法构建水稻育种知识图谱。水稻育种知识图谱的自顶向下构建流程如图2所示。
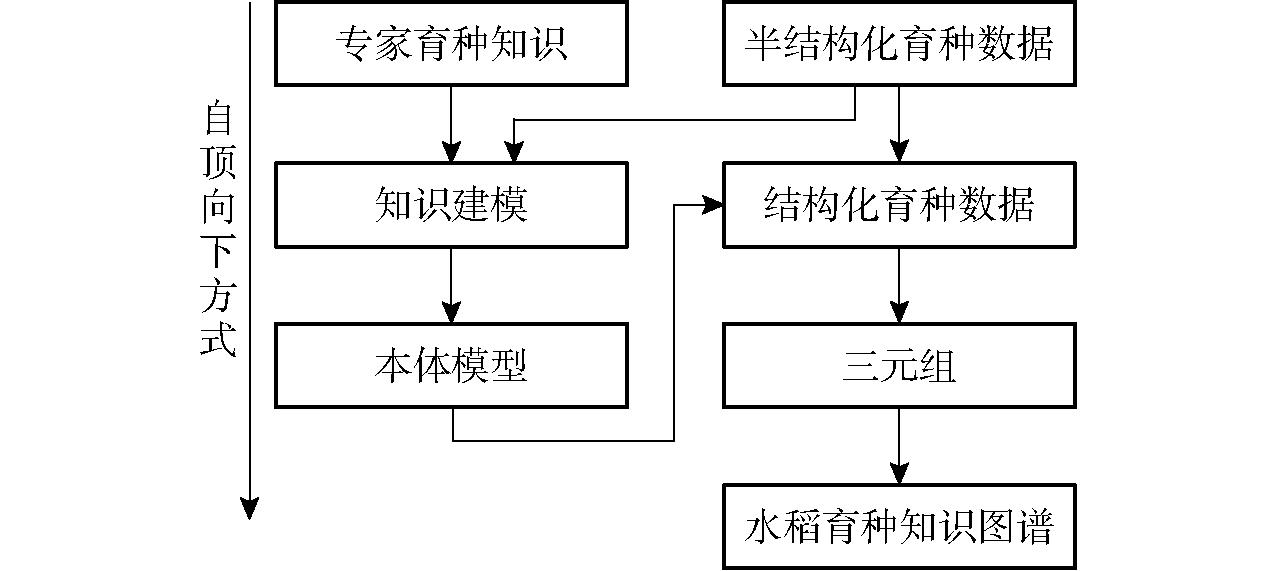
图2 水稻育种知识图谱自顶向下构建流程图
Fig.2 Top-down construction of rice breeding knowledge graph
由图2可知,首先根据专家育种知识和水稻育种数据进行知识建模,包括概念、属性以及关系,然后根据知识建模得到的本体模型去指导水稻育种文本中半结构化数据向结构化数据的转换,进一步抽取出结构化三元组。通过Cypher语言对三元组进行语义表示,并通过Neo4j图数据库实现三元组的导入,从而构建水稻育种知识图谱。水稻育种知识图谱构建的算法为:
输入:水稻育种数据Dr,水稻育种问句Qr
输出:水稻育种知识图谱Gr
1 Te←Qr的实体类型;
2 Td←Dr的文本类别;
3 根据Te和Td构建本体模型Or;
4 根据Te分类Dr的文本;
5 从Dr抽取结构化三元组TG;
6 批量导入TG到图数据库构成Gr;
其中水稻育种数据Dr以半结构化文本形式存储。以中国种业大数据平台为例,Dr以结构化(如品种来源、育种者等)和非结构化形式(如特征特性、栽培技术等)进行表示。根据水稻育种文本类别Td以及水稻育种问句中的实体类型Te,结合专家育种知识定义水稻育种本体模型Or,如图3所示。
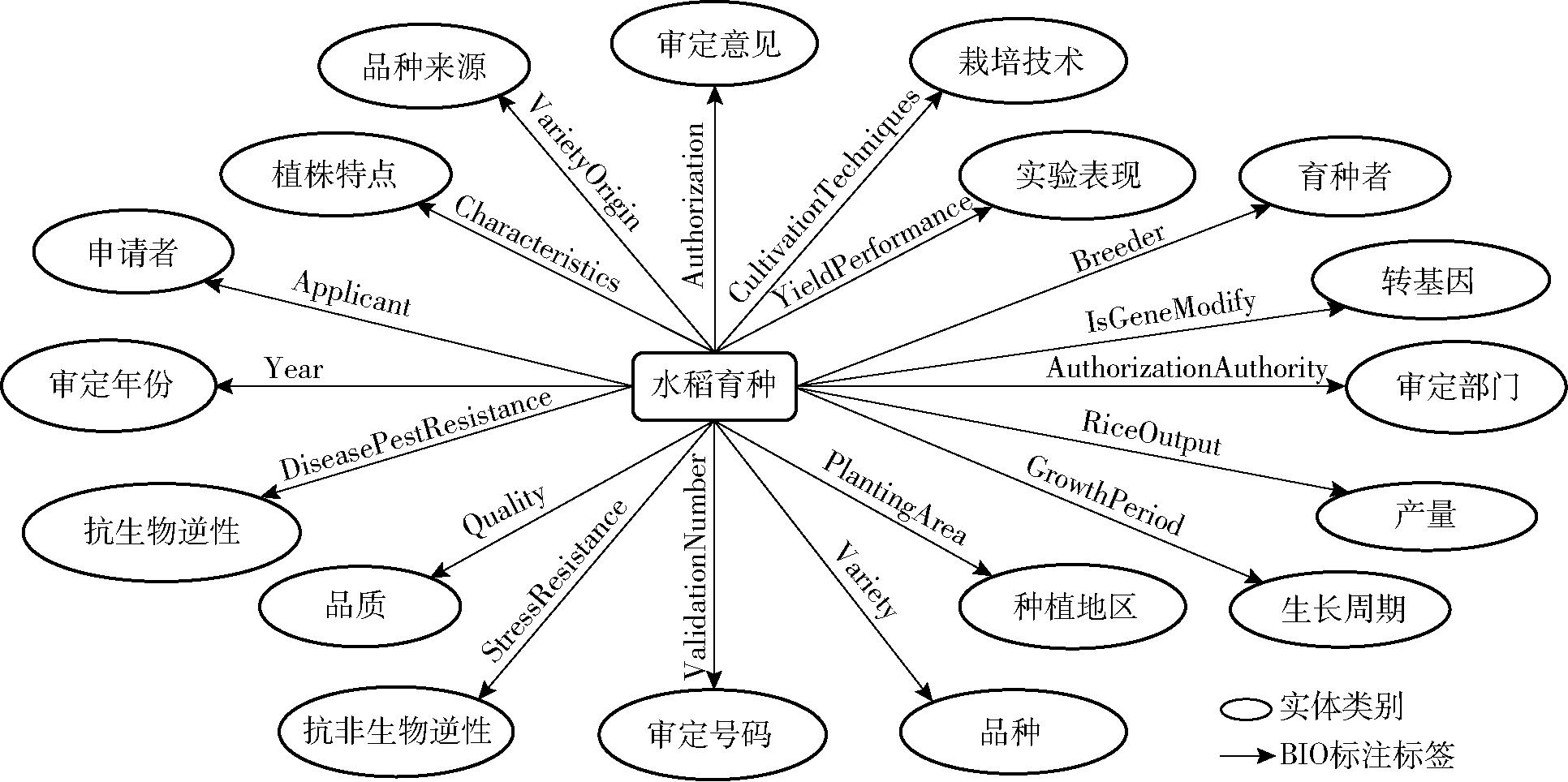
图3 水稻育种知识本体模型
Fig.3 Ontology model of rice breeding knowledge
考虑到Te,本文在Or中定义了与中文水稻育种问句Qr中命名实体相匹配的实体和实体属性。通过知识图谱的Or在原先大类实体上进一步划分出小类实体,如水稻特征特性类实体划分为抗生物逆性、生长周期、品质类、产量类、抗非生物逆性以及植株特点等小类实体。在半结构化的Dr文本中,相邻的以逗号分隔的句段,所属的实体属性相同而且为自然语言,进一步抽取会导致知识孤立。例如句段“出糙率80.8%~82.7%”,抽取后为80.8%和82.7%,难以表达该品种的出糙率正确的范围信息。针对该文本特点,本文采用基于规则的关键词匹配方法对Dr文本句段进行分类,从而降低文本信息的粒度。分类算法为:
输入: q={q1,q2,…,qN}, c={c1,c2,…,cM}
输出: R={R1,R2,…,RM}
1 μ←1;
2 While μ <N Do
3 j← 1;
4 While j <M Do
5 If keyword∈cjand keyword∈qμThen
6 Rj←Rj+qμ;
7 Else
8 continue;
9 End If
10 j← j+1;
11 End While
12 μ← μ+1;
13 End While
其中,N为句段总数,M为命名实体类别总数。q={q1,q2,…,qN}为Dr文本句段的集合。c={c1,c2,…,cM}为每类命名实体所属的关键词词典的集合。R={R1,R2,…,RM}为已经分类完成的不同类别句段的集合,即共M类的句段的集合。
通过每个命名实体类别的关键字词典与句段按序进行目标匹配,从而进行句段分类。例如,文本“适宜在黑龙江省第三积温带区域种植”匹配到水稻种植地区对应的命名实体关键词词典的一个关键词“区域种植”,根据本文中水稻种植地区对应的标签命名为“PlantingArea”,这一句段则分类到“PlantingArea”所属类别的句段集合。该方法有效提高了文本分类的效率。最后,从已经分类完成的Dr文本中抽取出结构化三元组TG(实体-关系-实体/实体属性)。将TG通过“LOAD CSV”语句批量导入Neo4j图数据库,构建水稻育种知识图谱Gr可视化结果如图4所示。通过构建Gr,可以提高水稻育种问答系统数据的管理水平,为Qr的命名实体类别划分提供参照,将Qr中的命名实体与Gr中的实体和实体属性相对应,可以作为实现水稻育种问答系统中基于实体进行检索信息的基础。
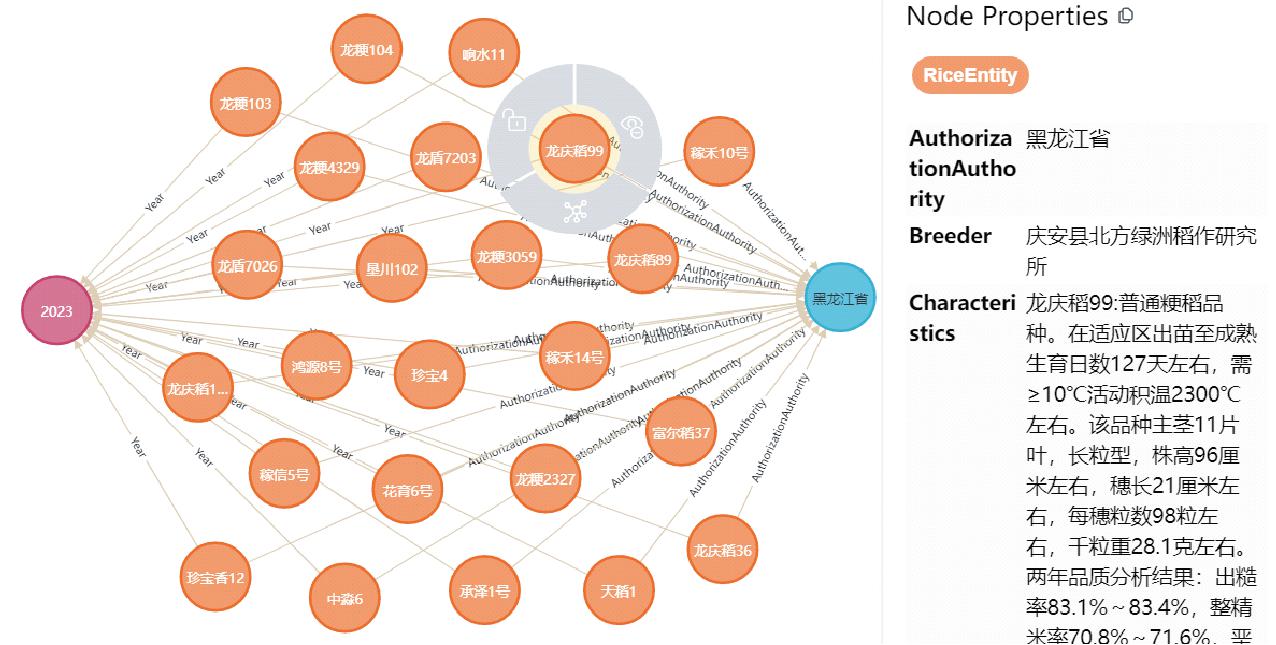
图4 水稻育种知识图谱可视化结果
Fig.4 Visual result of rice breeding knowledge
4 命名实体识别模型
DA层可以增强实体数目少的训练样本,BEAT预训练模型可以获取水稻育种问句动态词向量,BILSTM 层可以更好捕捉水稻育种连续问句间的依赖关系,CRF层可以在训练时自动学习到一些水稻育种命名实体的约束条件,减少错误预测序列。因此,本文提出了DA-BERT-BILSTM-CRF模型。需要训练的BIO标注问句文本输入到DA层进行增强,将增强的问句文本再输入到BERT层进行编码,输出句子的向量序列。BILSTM层将对这些序列进一步编码,将得到的每个类别的位置分数输入到CRF层计算最终的预测分数。所有类别的命名实体标注的标签均与水稻育种知识图谱中实体和实体属性相对应,详见图3。为了便于解释模型的输入和输出,以水稻育种问句文本中一个句段“粳稻的糙率”为例,来说明不同模型层的作用。其中“粳稻”实体属于水稻的品种类型(对应的命名为“Variety”),实验中标注的标签分别为“B-Variety”和“I-Variety”。“糙率”实体属于水稻的品质(对应的命名为“Quality”),标注的标签分别为“B-Quality”和“I-Quality”。其他非实体的无关字符标注为“O”。模型整体架构如图5所示。
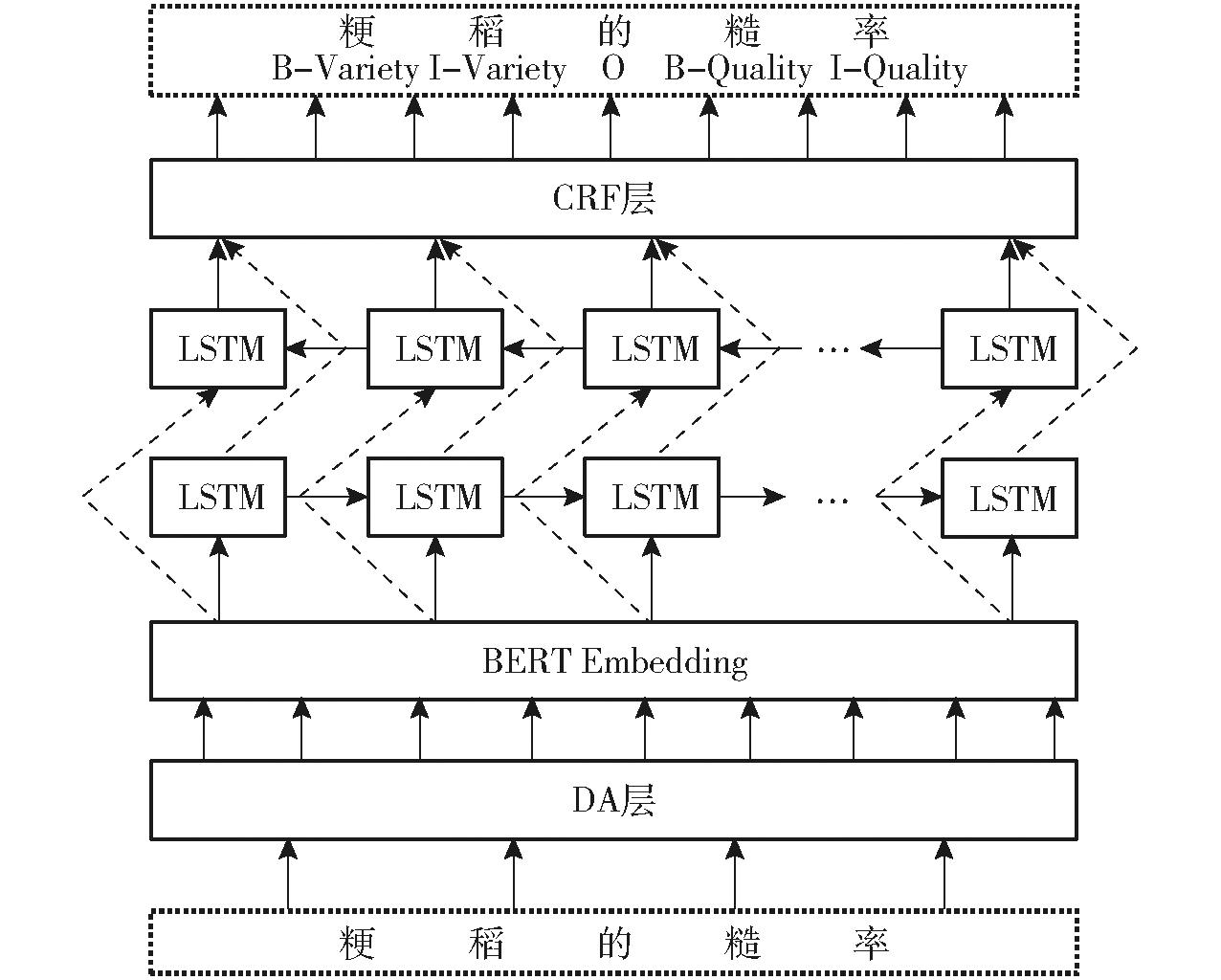
图5 命名实体识别模型架构
Fig.5 Architecture of proposed model
4.1 DA层
DA层可以增强训练数据以达到模型饱和训练。主流的DA技术有基于语言模型的生成方法DAGA、基于随机替换的实体增强方法EDA等。结构单一的水稻育种问句并不适用生成复杂结构语言文本的DAGA。而EDA技术实现代价小,效果接近基于深度学习的DA技术,不会改变句式结构,适用于水稻育种问句。EDA包括同义词替换、随机插入、随机交换以及随机删除。由于水稻育种数据中存在实体边界不一致、标注易混淆的问题,结合EDA和实例替换技术对DA层做了改进,根据水稻育种词汇在同标签实体替换时补充了一些边界清晰的实体信息,这使得增强后的数据文本噪声比例降低、边界清晰的实体比例提高,有助于提取不同类别实体的特征。改进的数据增强层DA结构如图6所示。图6中,![]() 为第i个类别水稻育种命名实体的第k个实体,i=1,2,…, M。Ni为第i类命名实体的实体总数。
为第i个类别水稻育种命名实体的第k个实体,i=1,2,…, M。Ni为第i类命名实体的实体总数。
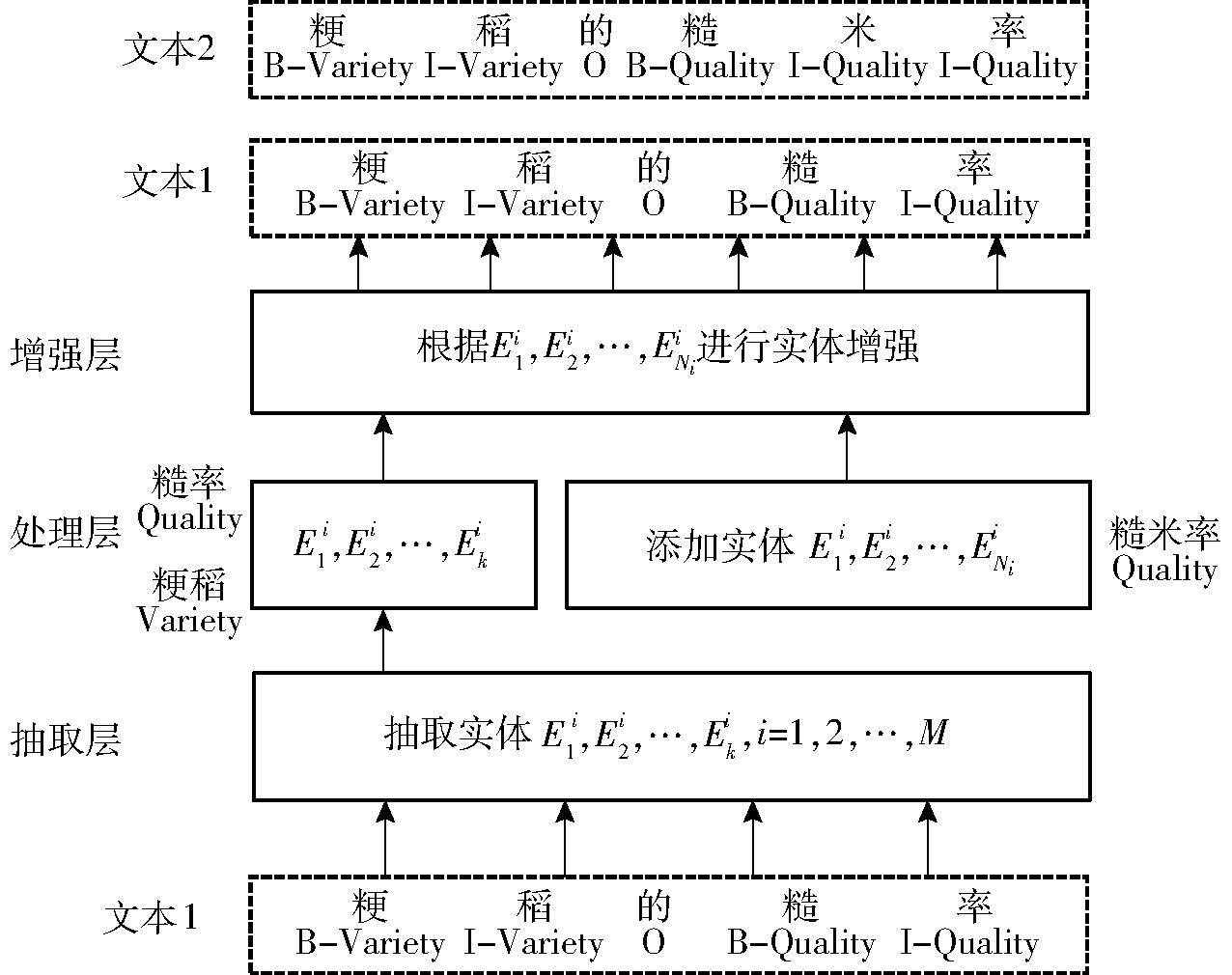
图6 数据增强层DA架构
Fig.6 Architecture of proposed DA layer
首先在抽取层将输入的BIO标注的问句抽取出![]() 然后在处理层与根据水稻育种词汇信息添加的
然后在处理层与根据水稻育种词汇信息添加的![]() 一起,作为第i类命名实体输入到增强层进行数据增强。这可以生成实体边界更清晰、特征更容易提取的BIO问句。以句段“粳稻的糙率”为例,输入到DA层的BIO标注序列为“B-Variety、I-Variety、O 、B-Quality、I-Quality”。通过抽取得到“Variety” 实体“粳稻”和“Quality”实体“糙率”。根据词汇信息添加的“Quality”实体“糙米率”,可以得到输出的增强句段“粳稻的糙米率”。DA层对标注数据进行增强可以降低边界不清晰或人工标注不准确的水稻育种实体比例,使得模型聚焦于提取更通用的实体特征,以提高模型的实体识别精确率。例如在图6中,对于输入DA层的标注文本1中出现的“糙率”一词,其作为标注后的实体并不符合正确的术语规范,而通过文本数据增强可以替换为具有正确实体边界的其他同类别实体,如输出文本2中的“糙米率”,以提高NER任务对该类别实体的识别性能。
一起,作为第i类命名实体输入到增强层进行数据增强。这可以生成实体边界更清晰、特征更容易提取的BIO问句。以句段“粳稻的糙率”为例,输入到DA层的BIO标注序列为“B-Variety、I-Variety、O 、B-Quality、I-Quality”。通过抽取得到“Variety” 实体“粳稻”和“Quality”实体“糙率”。根据词汇信息添加的“Quality”实体“糙米率”,可以得到输出的增强句段“粳稻的糙米率”。DA层对标注数据进行增强可以降低边界不清晰或人工标注不准确的水稻育种实体比例,使得模型聚焦于提取更通用的实体特征,以提高模型的实体识别精确率。例如在图6中,对于输入DA层的标注文本1中出现的“糙率”一词,其作为标注后的实体并不符合正确的术语规范,而通过文本数据增强可以替换为具有正确实体边界的其他同类别实体,如输出文本2中的“糙米率”,以提高NER任务对该类别实体的识别性能。
4.2 BERT层
BERT预训练语言模型使用了多层双向Transformer编码器,模型架构如图7所示。
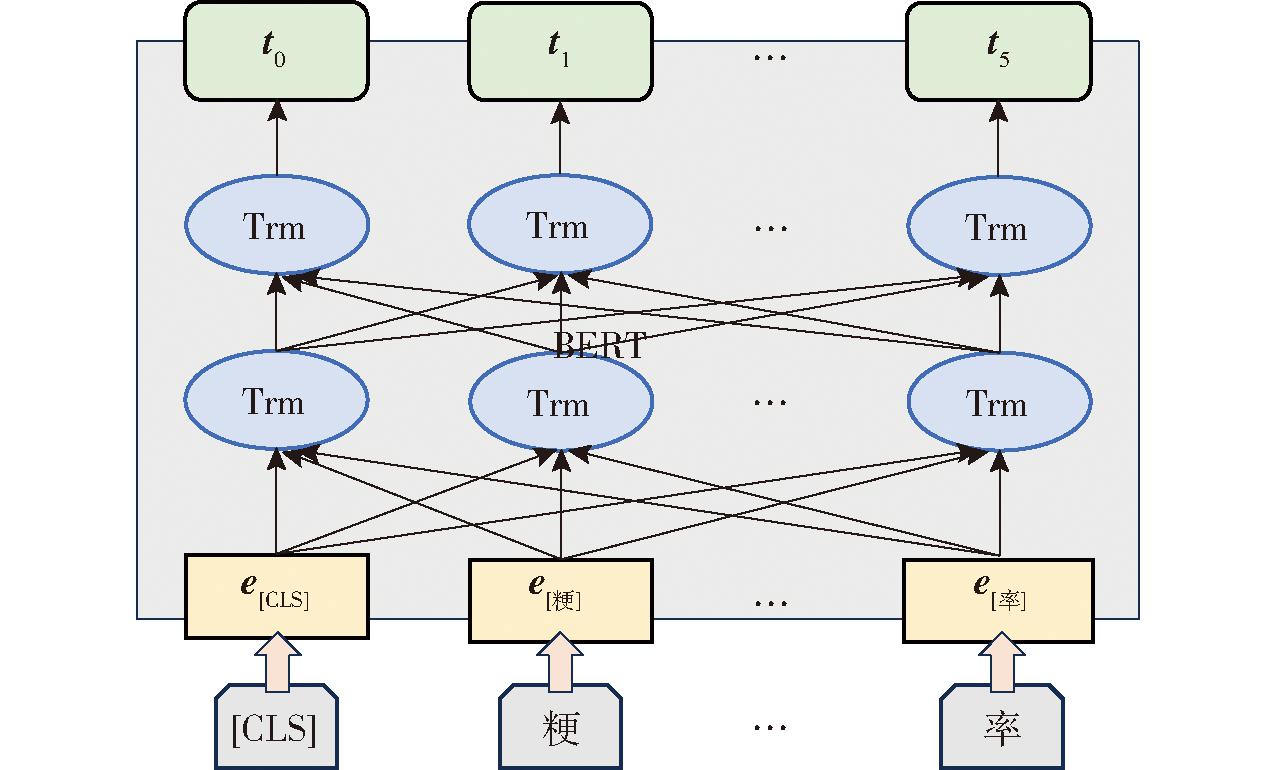
图7 BERT层模型架构
Fig.7 Architecture of BERT layer
BERT层可以根据一个水稻育种问句中单词之间的相关性来调整权重系数矩阵。以句段“粳稻的糙率”为例,BERT层把每个字转为对应的向量e,输入到模型中,经过双向Transformer(图7中简称为“Trm”)编码结构,输出每个字对应的融合整体语义信息的向量表示t1~t5。图7中在输入的文本前插入“[CLS]”符号,表示将输出向量作为整个文本的语义表示。相比于CNN,有着注意力机制的Transformer在自然语言处理领域有更好的实体特征提取能力,这说明BERT更适用于水稻育种实体特征的提取。由于BERT层从水稻育种文本中获取的动态词向量包含上下文联系,它可以更好地理解单词在水稻育种问句中的含义和整体的语义信息。有着大规模语料预训练的BERT模型也缓解了水稻育种NER任务对模型结构的依赖程度。
4.3 BILSTM层
BILSTM层由前向LSTM和后向LSTM组成,前向LSTM将负责生成水稻育种句段的隐藏状态序列,并由后向LSTM拼接这些隐藏状态序列,可以更好地捕捉水稻育种问句中双向的语义依赖。BILSTM可以学习到水稻育种句子上下文联系。动态词向量序列会输入到每个LSTM单元中,之后这些LSTM单元会学习提取出局部特征。LSTM单元的结构包含输入门、遗忘门和输出门。LSTM单元结构如图8所示。具体计算过程为
It=θ(WI[Ht-1,xt]+bI)
(1)
Ft=θ(WF[Ht-1,xt]+bF)
(2)
Ot=θ(WO[Ht-1,xt]+bO)
(3)
(4)
(5)
Ht=OtθT(Ct)
(6)
(7)
(8)
(9)
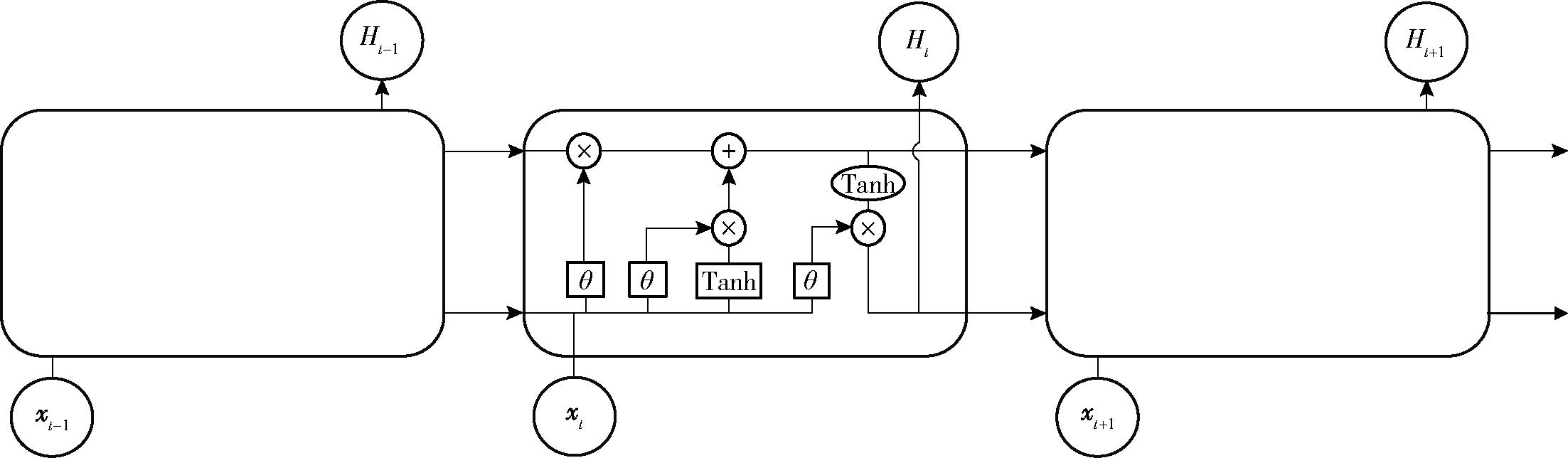
图8 LSTM单元结构
Fig.8 Architecture of LSTM unit
式中 It、Ft、Ot——LSTM计算单元的输入门、遗忘门、输出门
Ct——t时刻输出层输出状态
![]() 输出层状态候选者
输出层状态候选者
Ht——t时刻隐藏层输出状态
WI、WF、WO——对应门权重矩阵
xt——t时刻输入向量
bI、bF、bO——对应门LSTM偏差
Wc、bc——单元状态更新对应的权重矩阵、偏差
θ——激活函数
θT——双曲正切激活函数
L——LSTM计算单元输出函数
![]() t时刻前向LSTM和后向LSTM计算输出
t时刻前向LSTM和后向LSTM计算输出
![]() 和
和![]() 组成的输出
组成的输出
BILSTM架构图如图9所示。BILSTM层输出为水稻育种句段每个字对应的类别分数,并且将目前分数最高的类别作为输出的预测序列。然而仅BILSTM无法学习到标签之间的约束条件。例如图9中句段“粳稻的糙率”中“糙”字对应输入的字嵌入向量w3。由BILSTM层输出的w3对应不同标签位置分数的向量表示为(0.3, 0.2, 0.9, 1.3, 0.4),分别对应图9左边标签的向量表示为(B-Variety,I-Variety,B-Quality,I-Quality,O)。通过BILSTM层的输出结果选取最高分数1.3(图9中加下划线分数)的标签I-Quality与w3对应,即“糙”对应的预测标签为I-Quality。然而由于该I-Quality标签前面没有B-Quality,不符合BIO标注规则,这一预测结果是错误的。
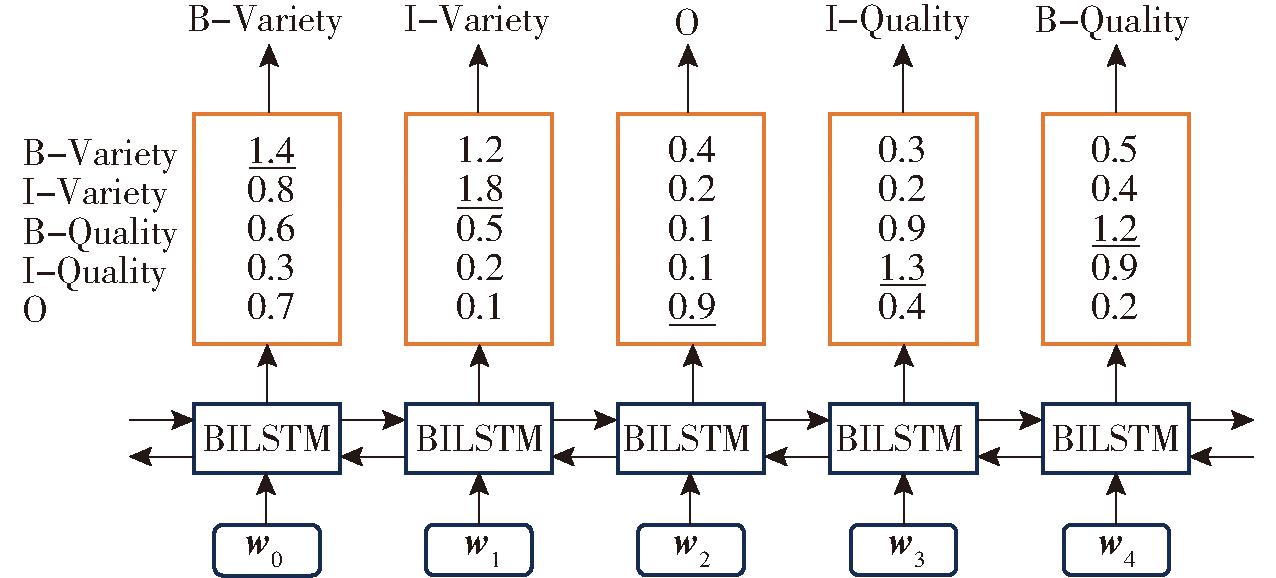
图9 BILSTM层架构
Fig.9 Architecture of BILSTM layer
4.4 CRF层
CRF层是由无向图组成的联合概率分布模型。作为最后一层,CRF层可以从水稻育种句段中学习到一些隐藏的约束条件来约束输出的预测标签。例如句段“粳稻的糙率”在预测“糙率”标签时,第1个I-Quality标签前应该有B-Quality标签等。CRF层可以保证输出的预测标签有效,解决了BILSTM层无法学习到标签之间的依赖关系问题。CRF层架构如图10所示。
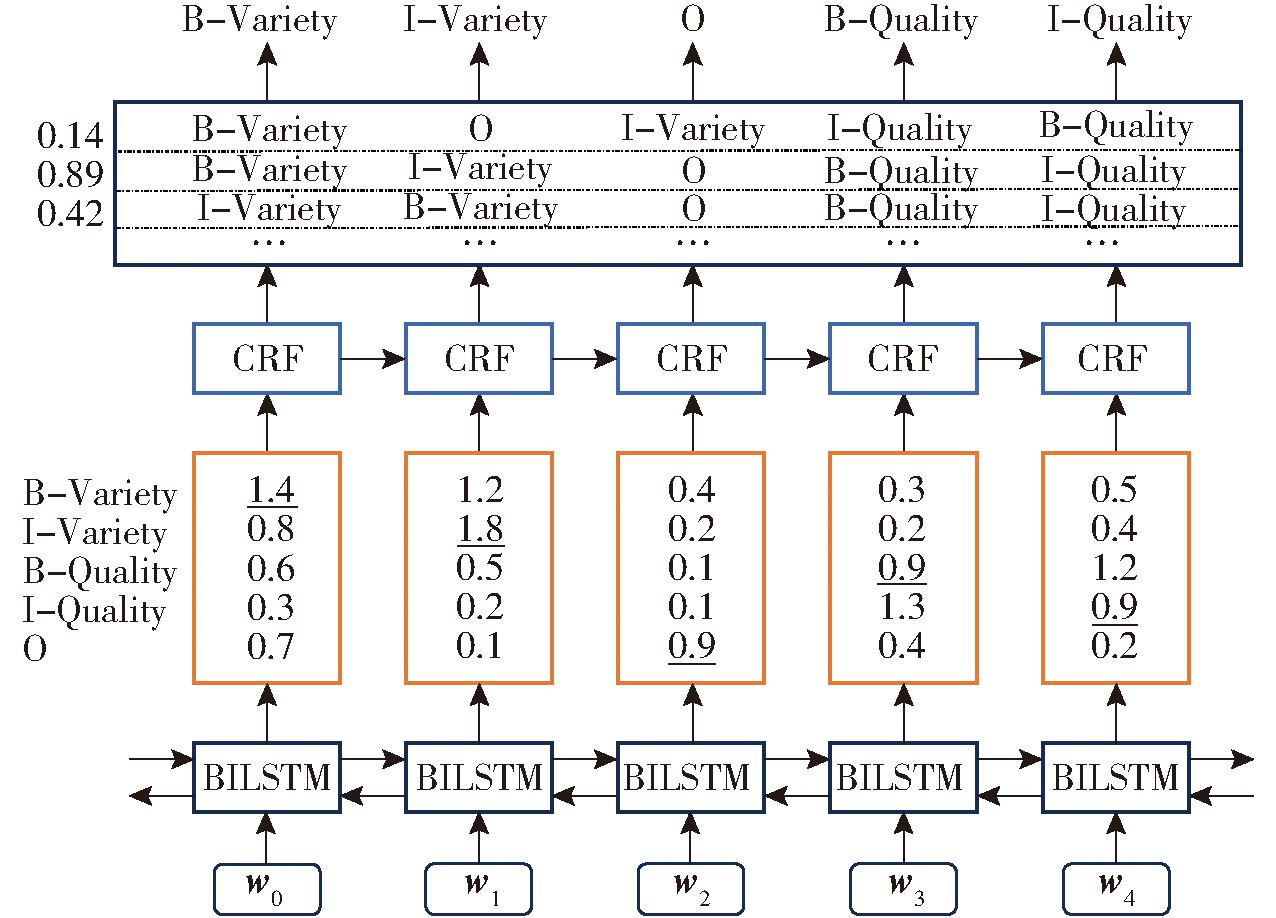
图10 CRF层架构
Fig.10 Architecture of CRF layer
由BILSTM层输出的水稻育种句段的分数将作为CRF层的输入。CRF层可以将局部特征归一化,计算整个输出序列的概率分布。标签位置概率分数计算公式为
(10)
式中 S——标签位置概率分数
X——水稻育种句段序列
Y——对应的模型标记序列概率
K——字的索引
YK——水稻育种标注标签的索引
PK,YK——对应的发射分数
AYK,YK+1——传递矩阵参数,表示对应的传递分数,即从YK标签移动到YK+1标签的概率
CRF层根据位置概率分数输出每个位置最高概率分数的预测标签,最后将计算出的分数最高标签序列作为水稻育种句段最终的预测结果。如图10中CRF层输出的句段“粳稻的糙率”的分数最高(0.89)的预测标注序列为B-Variety、I-Variety、O、B-Quality、I-Quality(图10中CRF层中加下划线序列)。对于单个字而言,与图9中的输出标签相比,CRF层不一定输出这个字在BILSTM层中最高分数对应的标签,而是输出这个字在分数最高的整体序列中对应的标签。例如“糙”字对应输入的字嵌入向量w3,通过BILSTM层输出的不同标签位置分数的向量表示同图9,为(0.3, 0.2, 0.9, 1.3, 0.4)。而通过CRF学习到的标签约束,最终w3对应的标签预测结果选取(0.3, 0.2, 0.9, 1.3, 0.4)中0.9(图10中BILSTM层加下划线分数代表模型最终输出选取的分数)对应的标签B-Quality,而不是分数最高(1.3)对应的标签I-Quality。加入CRF避免了模型输出水稻育种命名实体无效预测标签的问题。
5 实验与讨论
5.1 数据与参数设置
实验将以大类实体划分和小类实体划分2种情况分别对7 281个水稻用户问句数据样本进行标注,从而得到2种标注数据集。每次实验均随机划分出样本量5 000的训练集,验证集和测试集样本量均为1 000。实验选取的CPU为Intel(R) Core i9-13900HX,GPU为NVIDIA GeForce RTX 4060,内存为32 GB,硬盘容量为1 TB,编程语言选择Python 3.7,深度学习框架选取TensorFlow。鉴于输入单个水稻育种问句的字数量在个位数到两位数之间,所以选取的最大句长为128,实验通过验证集进行超参数调整,并经过多轮参数调整优化,将Gradient clip设置为5.0,Dropout设置为0.5,Batch-size设置为64,epoch为30,隐藏层单元数量为128,学习率为1×10-5。为清晰地说明实验中划分实体类别,水稻育种大类实体和小类实体的对照以及在数据集中的数量如表1所示。
表1 水稻育种实体对照
Tab.1 Comparison of rice breeding named entities
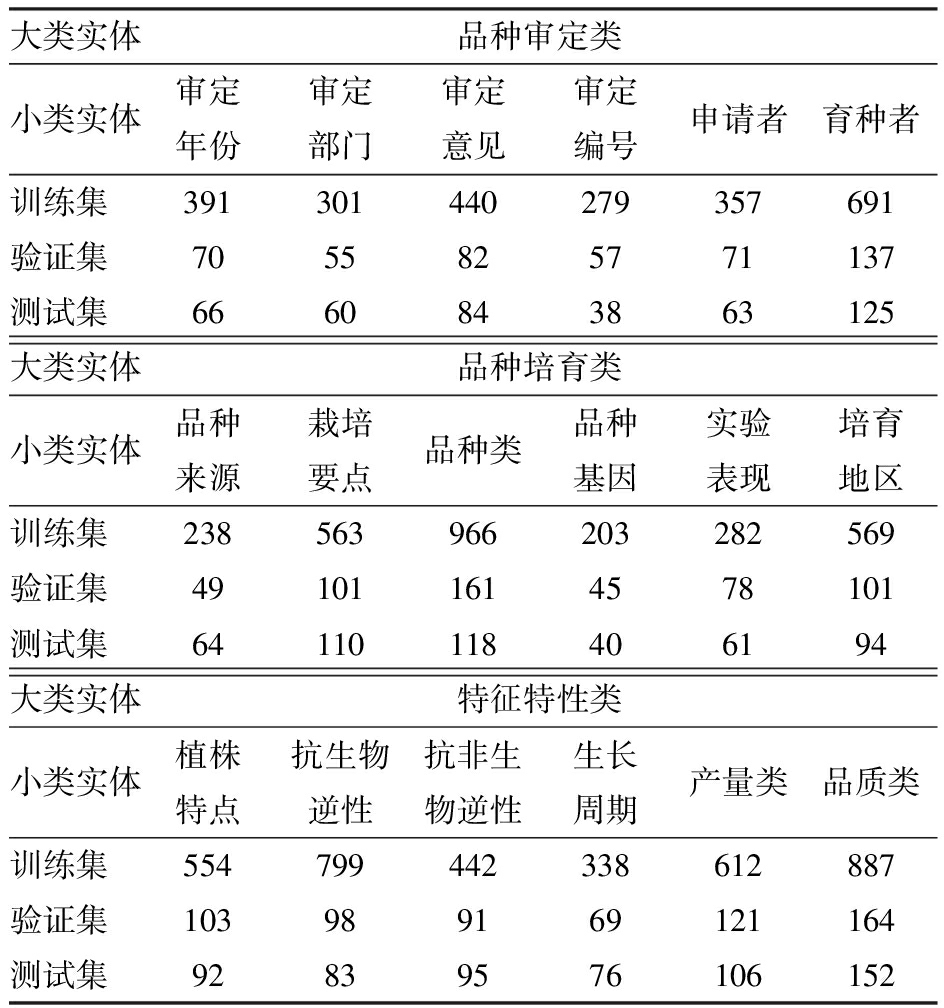
大类实体品种审定类小类实体审定年份审定部门审定意见审定编号申请者育种者训练集 391301440279357691验证集 7055825771137测试集 6660843863125大类实体品种培育类小类实体品种来源栽培要点品种类品种基因实验表现培育地区训练集 238563966203282569验证集 491011614578101测试集 64110118406194大类实体特征特性类小类实体植株特点抗生物逆性抗非生物逆性生长周期产量类品质类训练集 554799442338612887验证集 103989169121164测试集 92839576106152
5.2 实验评估指标
为评估模型性能,使用精确率(Precision)、召回率(Recall)以及F1值作为评估指标。实验将对模型在所有实体和单类别实体上分别进行评估。精确率用于评估模型识别正确实体的能力,召回率可以评估模型识别所有实体的能力,F1值则为精确率和召回率调和平均值,用于反映两者指标的实际平均情况。
5.3 原有实体划分下实验
为了验证所提出模型对水稻育种用户问句NER任务的有效性,实验将用该模型与其他基线模型BERT-CRF、BERT-BILSTM-CRF以及BILSTM-CRF进行对比。在水稻育种数据原有存储方式的大类实体的标注情况下,不同模型的水稻用户问句NER任务精确率、召回率、F1值对比如表2所示。
表2 不同模型在原有大类实体下的命名实体识别结果
Tab.2 NER results of different models under original large class entities %
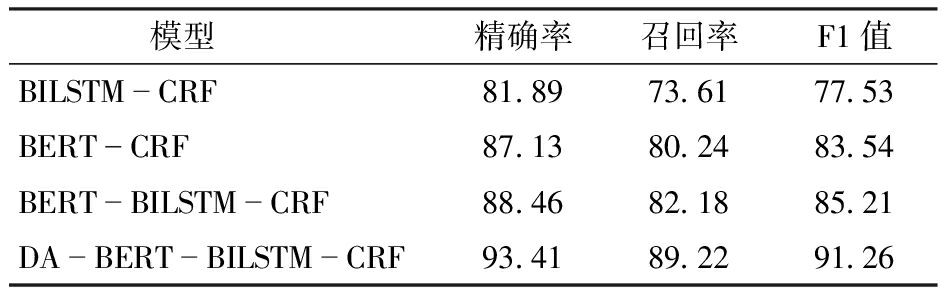
模型精确率召回率F1值BILSTMCRF81.8973.6177.53BERTCRF87.1380.2483.54BERTBILSTMCRF88.4682.1885.21DABERTBILSTMCRF93.4189.2291.26
从表2看出,4种模型整体命名实体的识别效果均较高,并且本文模型的识别效果更好。相比BILSTM-CRF和BERT-CRF,BERT-BILSTM-CRF识别精确率(88.46%)、召回率(82.18%)以及F1值(85.21%)均更高,原因在于,BERT层能获取全局的语义信息,可以更好地理解水稻育种文本,同时BILSTM层通过设置输出门、输入门以及遗忘门结构,能学习到水稻育种连续问句前后的联系。而本文模型通过改进的DA层使得它对实体特征的学习效果更好。从表2得出,在整体NER任务中本文模型精确率(93.41%)在数值上依次比BERT-BILSTM-CRF(88.46%)、BERT-CRF(87.13%)、BILSTM-CRF(81.89%)提高5.60%、7.21%、14.07%。DA-BERT-BILSTM-CRF模型F1值(91.26%)依次比BERT-BILSTM-CRF(85.21%)、BERT-CRF(83.54%)、BILSTM-CRF(77.53%)提高7.10%、9.24%、17.71%。然而由于知识粒度大和实体边界不明显,实验在进行每种实体识别时容易漏掉一些真实存在的实体,从而出现召回率低的问题。
5.4 小类实体划分下实验
在参照知识图谱将如水稻特征特性等大类实体划分出小类实体后,对这些实体分别进行标注,将提出模型与其他基线模型BERT-CRF、BERT-BILSTM-CRF、BILSTM-CRF进行对比。不同模型的水稻用户问句NER任务的指标对比如表3所示。
表3 不同模型在小类实体划分下的命名实体识别结果
Tab.3 NER results of different models under small class entity division %
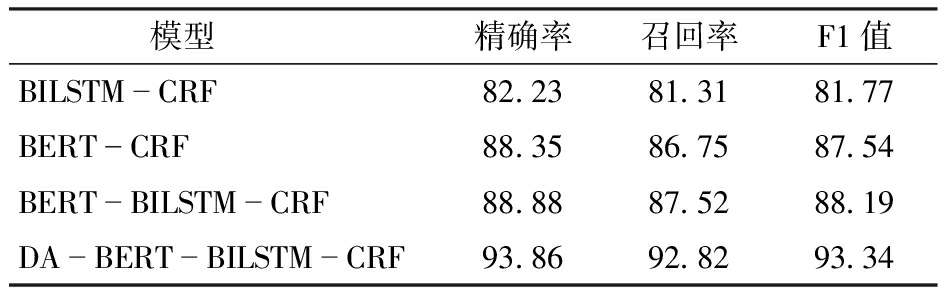
模型精确率召回率F1值BILSTMCRF82.2381.3181.77BERTCRF88.3586.7587.54BERTBILSTMCRF88.8887.5288.19DABERTBILSTMCRF93.8692.8293.34
表3中,BERT-BILSTM-CRF模型的精确率、召回率以及F1值在水稻育种问句NER任务实验的3个基线模型的指标中均有更好表现。例如,BERT-BILSTM-CRF模型的F1值(88.19%)分别比BERT-CRF(87.54%)、BILSTM-CRF(81.77%)提高0.74%和7.85%,同时这一对比说明BERT预训练模型在实体特征提取能力方面的表现对提高水稻育种NER任务性能有重要作用。其次,3个基线模型的召回率和精确率相差均在2个百分点以内,说明小类实体的实体边界更加清晰,词汇特征更容易识别,使得真实实体漏检的比率得到下降。而DA-BERT-BILSTM-CRF模型的精确率(93.86%)、召回率(92.82%)、F1值(93.34%)相比于BERT-BILSTM-CRF模型的精确率(88.88%)、召回率(87.52%)、F1值(88.19%)分别提高5.60%、6.06%、5.84%。这一对比结果说明,通过在DA层对标注文本数据增强时融入实体边界清晰的水稻育种词汇,模型能够更好地忽略不清晰或人工标注不准确的实体,从而提高了水稻育种问句NER任务的性能。
5.5 每类实体识别实验对比
在划分出小类水稻育种实体后,实验进一步统计出不同模型在识别每个类别命名实体的识别精确率如图11a所示;不同模型的每类命名实体的识别召回率如图11b所示;不同模型的每类命名实体的识别F1值如图11c所示。图11图例中的数值代表不同模型关于每类命名实体识别指标的平均值。
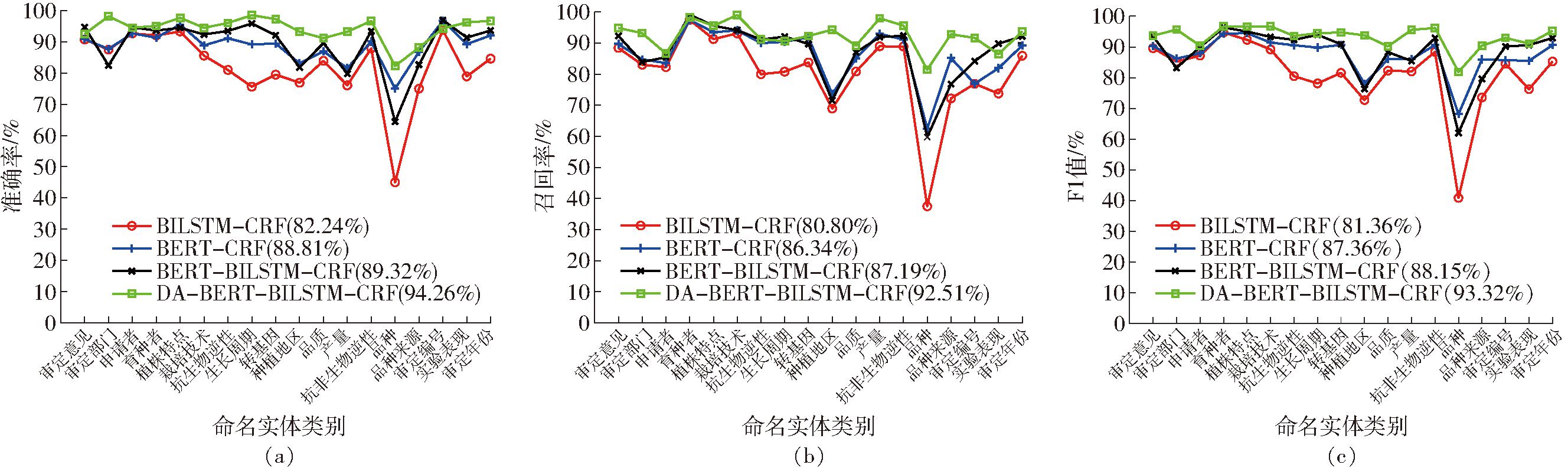
图11 单类别命名实体在不同模型下的识别性能对比
Fig.11 Comparison of recognition of each type of entity in different models
图11a中,BERT-BILSTM-CRF模型在3个基线模型中的表现最好,其单类别实体识别精确率达到89.32%,进一步验证了BERT层和BILSTM层对提高水稻育种实体识别精确率的有效性。而改进的DA层增强了标注文本数据,使得每种命名实体如品种类、种植地区等的特征更容易被模型聚焦学习,DA-BERT-BILSTM-CRF模型在单类别的实体识别任务的精确率表现相比于BERT-BILSTM-CRF模型更好,精确率均高于其他3种模型。同时单类别实体识别上,DA-BERT-BILSTM-CRF模型精确率(94.26%)相比于BERT-BILSTM-CRF(89.32%)、BERT-CRF(88.81%)、BILSTM-CRF(82.24%)分别提高5.53%、6.14%、14.62%。同样由图11b和图11c分析得到,DA-BERT-BILSTM-CRF模型在单类别实体识别的召回率和F1值的表现均优于其他模型,并且对于一些存在实体边界不一致和通用特征难学习等问题的命名实体如品种类等,通过文本数据增强,模型识别这些实体的召回率和F1值也均得到提高。
5.6 讨论
由表2和表3得出,4种模型在整体大类和小类实体识别任务中的精确率和F1值均较高。而将大类实体划分为小类实体,使得单类别命名实体边界更明显,通用特征更容易被模型学习,从而在表3 中,不同模型整体实体识别的效果相比于表2的效果均得到提高,同时降低了真实存在的实体漏检的比率,即假阴性实体的比率,显著提高了召回率。其中,DA-BERT-BILSTM-CRF的召回率(92.82%)和F1值(93.34%)相比于表2中的指标(89.22%,91.26%)分别提高3.60、2.08个百分点。这归功于在根据知识图谱的实体和实体属性划分出命名实体类别后,知识粒度降低,得到的小类命名实体的边界更加清晰,词汇特征更容易被提取。由此得出,在降低知识粒度和提高水稻育种命名实体任务性能的情况下,本文方法在整体命名实体的识别效果上更优。
在表2的大类实体标注数据集和表3的小类实体标注数据集中,4种模型在训练出的识别性能方面均表现良好。其中本文模型在2种数据集下均表现出更好的性能,水稻育种实体的识别精确率、召回率以及F1值均高于其他3种基线模型的对应指标。例如,DA-BERT-BILSTM-CRF模型在表2中的精确率为93.41%,高于其他3种模型精确率(88.46%、87.13%、81.89%),同时在表3中的精确率为93.86%,高于其他模型精确率(88.88%、88.35%、82.23%),因此DA-BERT-BILSTM-CRF模型具有较好的泛化能力。
从图11看出,DA-BERT-BILSTM-CRF模型在每个类别命名实体的识别精确率、召回率以及F1值上表现出的数值水平比较高且平均。对于一些容易造成问句意图识别不明、标注容易混淆以及实体边界不一致的实体类别如品种等,3个基线模型对这些类别的实体识别效果并不出色,例如对于品种类实体,BERT-BILSTM-CRF模型、BERT-CRF模型以及BILSTM-CRF模型识别F1值均不到70%。而融入水稻育种词汇信息的DA层可以使模型更容易学习到样本量小或标注边界不一致实体的特征,提高了模型对这一类实体的识别精确率、召回率以及F1值。例如在图11c中,DA-BERT-BILSTM-CRF模型在品种类、种植地区等实体上识别的F1值明显高于其他基线模型对应的F1值。
DA-BERT-BILSTM-CRF在表2和表3中的整体实体识别效果均更优,表现出了文本数据增强对提高水稻育种问句NER任务性能的有效性。同时由表2和表3的实验对比得出,参照知识图谱划分出小类命名实体类别,使得水稻育种实体边界更加清晰,知识粒度降低,并提高了DA层对水稻育种实体进行增强的效果。之后由图11结果分析得到,所提出方法在单类别的水稻育种实体识别任务的精确率、召回率以及F1值均有更高的水平。综合上述实验分析得出,本文方法在水稻育种问句NER任务中有更好的性能表现,具有可行性。
目前在基于规则的水稻育种文本的划分工作中会出现冗余情况,并且由于资源有限,结合不同标注方式只验证了此方法在2种数据集下的泛化能力,之后将寻求其他水稻育种标注数据进一步验证泛化性。
6 结束语
针对水稻育种问答应用数据管理水平不高以及缺乏标注数据等问题,提出了一种基于文本数据增强的水稻育种问句命名实体识别方法。相比于现有技术,所提出方法结合EDA和实例替换技术,使得增强文本更加多样;在增强的实体中融入水稻育种词汇,提高了边界清晰的实体比例,使得模型聚焦于每类实体的通用特征;参照知识图谱将水稻育种大类实体划分出小类实体,提高了文本数据增强的效果。实验表明,本文方法优于其他方法,其中在单类别识别精确率达到94.26%,F1值达到93.32%,分别比现有方法提高5.53%和5.87%;整体识别任务精确率达到93.86%,F1值达到93.34%,分别比现有方法提高5.60%和5.84%。
[1] BAI Shiwei, YU Hong, WANG Bing, et al. Retrospective and perspective of rice breeding in China[J]. Journal of Genetics and Genomics,2018,45(11):603-612.
[2] LI Jiming, LUO Xiaohe, ZHOU Kunlu. Research and development of hybrid rice in China[J]. Plant Breeding,2023,1(1):1-9.
[3] 梁敬东,崔丙剑,姜海燕,等.基于Word2vec和LSTM的句子相似度计算及其在水稻FAQ问答系统中的应用[J].南京农业大学学报,2018,41(5):946-953.
LIANG Jingdong, CUI Bingjian, JIANG Haiyan, et al. Sentence similarity computing based on Word2vec and LSTM and its application in rice FAQ question-answering system[J]. Journal of Nanjing Agricultural University,2018,41(5):946-953. (in Chinese)
[4] WANG Haoriqin, WU Huarui, ZHU Huaji, et al. Aresidual LSTM and Seq2Seq neural network based on GPT for Chinese rice-related question and answer system[J]. Agriculture,2022,12(6):813.
[5] 陶星星,吴亚辉,付魏魏,等.水稻育种信息数据管理系统的设计与开发[J].中国种业,2019,1(6):4-7.
TAO Xingxing, WU Yahui, FU Weiwei, et al. Design and development of rice breeding information data management system[J]. China Seed Industry,2019,1(6):4-7. (in Chinese)
[6] 赖英旭,李亚娟,刘静.基于本体的水稻育种方法应用知识库构建[J].北京工业大学学报,2019,45(12):1181-1191.
LAI Yingxu, LI Yajuan, LIU Jing. Construction of ontology-based rice breeding method knowledge base[J]. Journal of Beijing University of Technology,2019,45(12):1181-1191. (in Chinese)
[7] 袁培森,李润隆,王翀,等.基于BERT的水稻表型知识图谱实体关系抽取研究[J].农业机械学报,2021,52(5):151-158.
YUAN Peisen, LI Runlong, WANG Chong, et al. Entity relationship extraction from rice phenotype knowledge graph based on BERT[J]. Transactions of the Chinese Society for Agricultural Machinery,2021,52(5):151-158. (in Chinese)
[8] 侯琛,牛培宇.农业知识图谱技术研究现状与展望[J]. 农业机械学报,2024,55(6):1-17.
HOU Chen, NIU Peiyu. Review of research status and prospects of agricultural knowledge graphs[J]. Transactions of the Chinese Society for Agricultural Machinery, 2024,55(6):1-17. (in Chinese)
[9] STEINER T, VERBORGH R, TRONCY R, et al. Adding realtime coverage to the Google knowledge graph[C]∥11th International Semantic Web Conference, 2012:65-68.
[10] YAN Jihong, WANG Chengyu, CHENG Wenliang, et al. A retrospective of knowledge graphs[J]. Frontiers of Computer Science,2018,12(1):55-74.
[11] CHEN Xiaojun, JIA Shengbin, XIANG Yang. A review: knowledge reasoning over knowledge graph[J]. Expert Systems with Applications,2020,141(1):112948.
[12] 徐增林,盛泳潘,贺丽荣,等.知识图谱技术综述[J].电子科技大学学报,2016,45(4):589-606.
XU Zenglin, SHENG Yongpan, HE Lirong, et al. Review on knowledge graph techniques[J]. Journal of University of Electronic Science and Technology of China,2016,45(4):589-606. (in Chinese)
[13] PENG Ciyuan, XIA Feng, NASERIPARSA M, et al. Knowledge graphs: opportunities and challenges[J]. Artificial Intelligence Review,2023,56(11):13071-13102.
[14] LIN Jinjiao, ZHAO Yanze, HUANG Weiyuan, et al. Domain knowledge graph-based research progress of knowledge representation[J]. Neural Computing and Applications,2021,33(2):681-690.
[15] 于合龙,沈金梦,毕春光,等.基于知识图谱的水稻病虫害智能诊断系统[J].华南农业大学学报,2021,42(5):105-116.
YU Helong, SHEN Jinmeng, BI Chunguang, et al. Intelligent diagnostic system for rice diseases and pests based on knowledge graph[J]. Journal of South China Agricultural University,2021,42(5):105-116. (in Chinese)
[16] 戈为溪,周俊,袁立存,等.基于知识图谱与案例推理的水稻精准施肥推荐模型[J].农业工程学报,2023,39(2):126-133.
GE Weixi, ZHOU Jun, YUAN Licun, et al. Recommendation model for rice precision fertilization using knowledge graph and case-based reasoning[J]. Transactions of the CSAE,2023,39(2):126-133. (in Chinese)
[17] DING Bosheng, LIU Linlin, BING Lidong, et al. DAGA: data augmentation with a generation approach for low-resource tagging tasks[C]∥Proceedings of the 2020 Conference on Empirical Methods in Natural Language Processing, Online,2020:6045-6057.
[18] WEI Jason, ZOU Kai. EDA: easy data augmentation techniques for boosting performance on text classification tasks[C]∥Proceedings of the 2019 Conference on Empirical Methods in Natural Language Processing and the 9th International Joint Conference on Natural Language Processing, 2019:6382-6388.
[19] KIM J, KIM Y, KANG S. Weakly labeled data augmentation for social media named entity recognition[J]. Expert Systems with Applications,2022,209(1):118217.
[20] BARTOLINI I, MOSCATO V, POSTIGLIONE M, et al. COSINER: context similarity data augmentation for named entity recognition[C]∥International Conference on Similarity Search and Applications, 2022:11-24.
[21] TIKHOMIROV M, LOUKACHEVITCH N, SIROTINA A, et al. Using BERT and augmentation in named entity recognition for cybersecurity domain[C]∥International Conference on Applications of Natural Language to Information Systems,2020:16-24.
[22] 李想,魏小红,贾璐,等.基于条件随机场的农作物病虫害及农药命名实体识别[J].农业机械学报,2017,48(增刊):178-185.
LI Xiang, WEI Xiaohong, JIA Lu, et al. Recognition of crops, diseases and pesticides named entities in Chinese based on conditional random fields[J]. Transactions of the Chinese Society for Agricultural Machinery,2017,48(Supp.):178-185. (in Chinese)
[23] 刘新亮,张梦琪,谷情,等.基于BERT-CRF模型的生鲜蛋供应链命名实体识别[J].农业机械学报,2021,52(增刊):519-525.
LIU Xinliang, ZHANG Mengqi, GU Qing, et al. Named entity recognition of fresh egg supply chain based on BERT-CRF architecture[J]. Transactions of the Chinese Society for Agricultural Machinery,2021,52(Supp.):519-525. (in Chinese)
[24] 李林,周晗,郭旭超,等.基于多源信息融合的中文农作物病虫害命名实体识别[J].农业机械学报,2021,52(12):253-263.
LI Lin, ZHOU Han, GUO Xuchao, et al. Named entity recognition of diseases and insect pests based on multi source information fusion[J]. Transactions of the Chinese Society for Agricultural Machinery,2021,52(12):253-263. (in Chinese)
[25] DEVLIN J, CHANG Mingwei, LEE K, et al. BERT: pre-training of deep bidirectional transformers for language understanding[C]∥Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies,2019:4171-4186.
[26] PETERS M E, NEUMANN M, IYYER M, et al. Deep contextualized word representations[C]∥Proceedings of the 2018 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies,2018:2227-2237.
[27] ZHANG Wentao, JIANG Shaohua, ZHAO Shan, et al. A BERT-BiLSTM-CRF model for Chinese electronic medical records named entity recognition[C]∥2019 12th International Conference on Intelligent Computation Technology and Automation,2019:166-169.
[28] MENG Fanqi, YANG Shuaisong, WANG Jingdong, et al. Creating knowledge graph of electric power equipment faults based on BERT-BiLSTM-CRF model[J]. Journal of Electrical Engineering &Technology,2022,17(4):2507-2516.