奶粉的质量安全问题一直是消费者关注的焦点。然而由于商业利益驱使,且掺伪手段容易且较难检出,使得奶粉成为食品掺假行为频发领域,并已成为全球性食品安全问题[1]。
目前,奶粉掺假检测采用随机抽样、实验室送检方式,流程长效率低。多数借助于化学方法,主要包括高效液相色谱法[2]、气相色谱法[3]、质谱法[4]、毛细管电泳法[5]和荧光光谱法[6]等。然而,这些常规方法往往需要专业、繁琐的前处理步骤和复杂、昂贵的分析仪器,导致其单次样品检测时间长,难以满足现场快速实时评价的检测需求。近年来,光谱技术快速发展,其无损、快速和绿色等检测优点,为乳粉掺杂检测提供了一种高效的新手段。其中,高光谱成像技术基于含氢基团振动吸收和二维空间成像的原理,能通过光谱和图像,获取内部成分和外部物理特征信息,在肉类[7]、农产品(如水稻[8]、玉米[9]、大米[10])和调味品(如香料[11])等的掺假检测领域均取得了系列研究成果。
对于奶粉掺假检测,文献[12]通过高光谱成像结合PLS(偏最小二乘)算法,在990~1 700 nm光谱范围内检测奶粉中掺杂的三聚氰胺,基于可视化PLS结果图和阈值法能够检测混合样本中低至0.02%掺假质量分数水平的三聚氰胺颗粒。文献[13]通过近红外高光谱成像技术和多元曲线分辨率(Multivariate curve resolution,MCR)方法对奶粉一元(乳清粉、淀粉、尿素或三聚氰胺)、二元(淀粉和尿素)和三元(淀粉、尿素和乳清粉)等不同复杂情况下的掺假检测进行了研究。所提出的方法不需要大量的先验信息,预测的掺假物质量分数的绝对误差均低于5%。以上研究表明高光谱成像技术结合适当的分析算法,可以有效检测奶粉中低含量和多元掺假情况。
高光谱图像数据的共线性、冗余性和高维性,以及近红外谱区吸收峰重叠等特点,使其数据解析和特征挖掘成为一项艰巨的任务。不同于以往的建模算法,深度学习算法基于端到端的建模思想,从输入数据中自动提取抽象特征并学习,预测结果可达到较理想的精确水平。文献[14]利用近红外光谱结合基于注意力机制的集成卷积神经网络(Convolutional neural networks,CNN)建模方法,检测不同品牌婴幼儿配方奶粉中单一掺杂的水解皮革蛋白和三聚氰胺,与经典回归方法(PLS、核偏最小二乘(Kernel partial least squares,K-PLS)和CNN)相比,所提出的集成方法表现出更优越的回归性能。
尽管已有以上较多针对掺假奶粉的检测研究,但大多主要讨论单一掺假情况,且奶粉样本的品牌类型较单一,而实际掺假情况往往比较复杂。本文选取4种品牌的婴幼儿配方奶粉,向其中同时掺入三聚氰胺、香兰素和淀粉3种掺假物,分别建立基于特征选择算法的经典机器学习模型和深度学习卷积神经网络模型。利用两种经典过滤式特征选择算法:无监督的拉普拉斯分数(Laplacian score)和有监督的ReliefF,建立PLS模型。构建能进行解释性分析的添加自定义选择层的1DCNN模型[15],对不同掺假物进行定量预测。讨论3种方法确定的最优波长并对最优模型的预测结果进行可视化分析。
从河北省保定市某大型超市购买4个品牌的1段(0~6个月)婴幼儿配方奶粉:A(飞鹤)、B(完达山)、C(贝因美)、D(雅培)。香兰素和三聚氰胺购自北京易秀博谷生物科技有限公司,纯度99.5%以上。按照表1所示,将奶粉与不同质量分数的三聚氰胺、香兰素和淀粉充分混合,制备多掺假样品。
表1 掺假样品中各掺杂物的混合质量分数
Tab.1 Mass fractions of different adulterants in mixed samples %
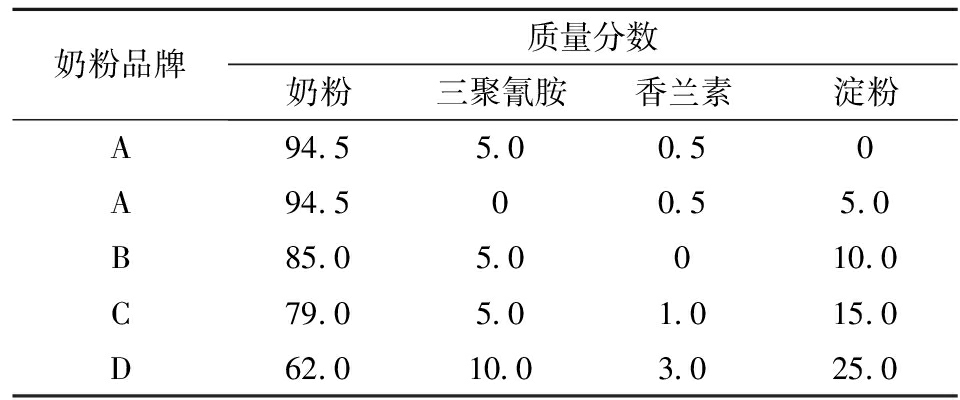
奶粉品牌质量分数奶粉三聚氰胺香兰素淀粉A94.55.00.50A94.500.55.0B85.05.0010.0C79.05.01.015.0D62.010.03.025.0
将每种混合物放入3个塑料方形培养皿(100 mm×100 mm×15 mm)中,粉末层厚度约占培养皿高度的1/2。按照相同方式制备纯奶粉、三聚氰胺、香兰素和淀粉样本。
为了验证文中所建方法对外部验证样品的预测能力,从网上购买同样4个品牌的不同批次奶粉样品。三聚氰胺、香兰素购自上海麦克林生化科技有限公司,淀粉购自国药集团化学试剂有限公司。同样按照表1所示质量分数制备混合物样品作为外部验证集。此外,为了进一步分析模型对不同品牌奶粉掺假的预测能力,如表2所示,设计了新的外部验证样品。新外部验证样本制备所用的奶粉和掺假物与外部验证样品所用相同,每种混合物放于一个塑料方形培养皿进行后续的数据采集。
表2 新外部验证集混合掺假样品的奶粉品牌和各物质质量分数
Tab.2 Milk powder brands and mass fractions of different adulterants of mixed adulterated samples in new external validation set %
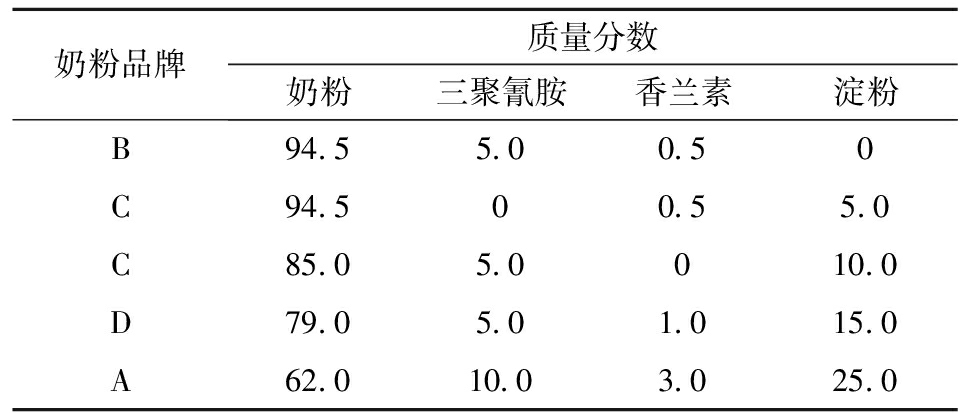
奶粉品牌质量分数奶粉三聚氰胺香兰素淀粉B94.55.00.50C94.500.55.0C85.05.0010.0D79.05.01.015.0A62.010.03.025.0
使用推扫式高光谱成像系统(SPECIM SisuCHEMA型,Spectral Imaging Itd.公司,芬兰)(图1)采集样品的高光谱图像。基于InGaAs检测器的线扫描高光谱相机(SPECIM FX10型,Spectral Imaging Ltd.公司,芬兰)采集的光谱范围为935.61~1 720.23 nm,光谱分辨率为3.45 nm,图像分辨率约为0.26 mm/像素。
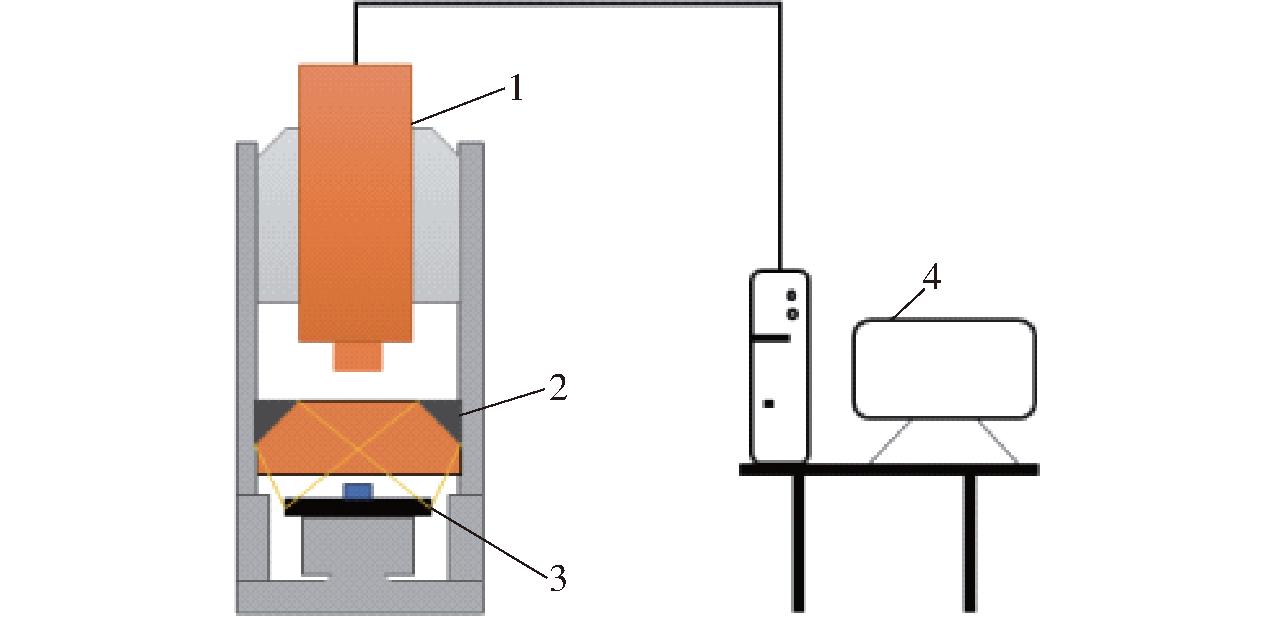
图1 高光谱成像仪结构示意图
Fig.1 Schematic of hyperspectral imaging system
1.高光谱成像仪 2.光源 3.载物台 4.计算机
为了减少暗电流和环境光变化等因素对高光谱图像的影响,需要对原始高光谱图像进行黑白校正。校正公式为
式中 R——校正图像反射率
l0——原始图像反射强度
B——黑参考图像反射强度
W——白参考图像反射强度
为了去除每幅图像中培养皿和背景等不相关像素,选择3个培养皿中的粉末像素作为感兴趣区域(ROI),并从校正图像中裁剪出来。每个裁剪子图像尺寸为360(像素)×360(线)×224(波段)。总共获得15个子图像(5个含量梯度×3个重复)。粉末样品反射率在光谱范围936~1 157 nm内几乎饱和(接近100%)。此外,1 700 nm之后光谱范围内的数据通常信噪比较低。因此,研究仅对1 158~1 702 nm的155个波段数据进行分析和处理。
1.3.1 预处理
为了消除探测器灵敏度差异、样本不规则表面散射等干扰对光谱数据的不利影响,将亮度校正、吸收光谱转换和标准正态变换(Standard normal variate,SNV)等预处理算法应用于高光谱图像中的每个像素点光谱,具体说明详见文献[16]。
随后,每个子图像被均等分成9个ROI,并计算ROI平均光谱。每种粉末混合物和纯粉末样品,提取27个ROI平均光谱(9个ROI×3个平行),共获得135条掺假样本光谱(27条光谱×5个掺假比例)。
1.3.2 偏最小二乘回归
偏最小二乘是一种经典的定量建模方法,它通过将大量自变量减少为一组新的不相关变量(称为潜在变量)来避免多重共线[17]。
1.3.3 卷积神经网络
CNN通过正向和反向传播更新神经网络的权值,以最小化预测值与真实标签之间的差异。本文设计了一种卷积神经网络模型,包括1层输入层、1层自定义层、2层卷积层、2层激活层、2层池化层、1层全连接层和1层输出层。网络学习率和迭代次数分别为0.001和700。
1.3.4 特征波长选择
特征选择(Feature selection,FS)[18]是解决高维数据维度灾难问题的主要降维方法。过滤式特征选择算法具有独立于训练模型、结构简单、速度快的优点。本文采用了两种经典的过滤式特征选择方法,分别是无监督的拉普拉斯分数(Laplacian score)和有监督的ReliefF。Laplacian score基于局部保持投影和拉普拉斯特征映射,结合局部结构信息和方差,根据特征的局部保持能力来评估特征得分[19]。ReliefF基于特征对近距离样本的区分能力度量特征与类别标签的相关性,基于此计算特征的重要性[20]。
1.3.5 预测结果可视化
从粉末混合物中随机选择尺寸为150像素×150像素的ROI用于可视化。将感兴趣区域中每个像素点视作一个样本提取光谱构建二维(2-D)矩阵输入最优模型,获得可视化预测结果。
图2为纯样本奶粉和3种掺假物:三聚氰胺、香兰素和淀粉的平均光谱。4种物质的光谱曲线趋势总体差异明显,为以后的分类预测提供了基础。掺假物中淀粉与奶粉的光谱曲线趋势相近,但奶粉在1 210 nm处的吸收峰更高,该吸收峰与蛋白质中C—H键伸缩振动的第二泛音有关[21]。香兰素、奶粉和淀粉均在1 577 nm处出现吸收峰,其中香兰素在该处的吸收较强,奶粉和淀粉该处吸收较弱。三聚氰胺在1 524 nm和1 492 nm处存在的2个较强吸收峰,分别与N—H对称和反对称拉伸振动的第一泛音有关,与文献[22]研究结果一致。三聚氰胺中芳香胺结构的存在,使得三聚氰胺和其他物质在 1 467 nm左右的光谱处存在最显著的光谱差异[23]。
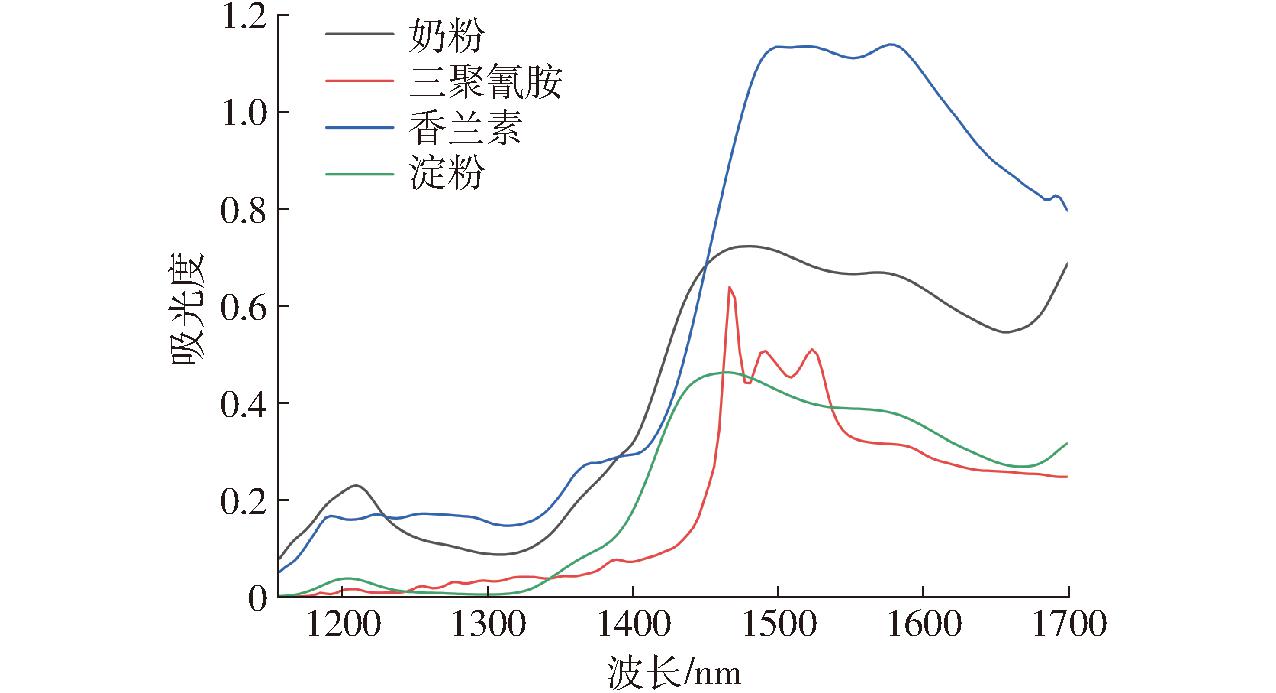
图2 奶粉、三聚氰胺、香兰素和淀粉纯样品的平均光谱
Fig.2 Average spectra of pure samples of milk,melamine,vanillin and starch
2.2.1 全波段PLS模型
将每种混合类型的27条光谱按照比例2∶1随机划分为校正集和预测集,共得到校正集90条光谱(18条×5种),预测集45条光谱(9条×5种)。采用五折法建立交叉验证集。通过交叉验证均方根误差(RMSEV),确定最优潜变量因子数。PLS全波长模型预测结果如表3所示。可以看出,奶粉、三聚氰胺、香兰素和淀粉预测模型的校正集、交叉验证集和预测集决定系数R2均大于0.960 0,表明建立的PLS模型能够有效预测混合物中各物质含量。根据交叉验证集均方根误差,模型对三聚氰胺的预测效果最优,淀粉次之,其次是奶粉,香兰素最差。
表3 PLS全波长模型预测结果
Tab.3 Prediction results of PLS models based on full wavelengths
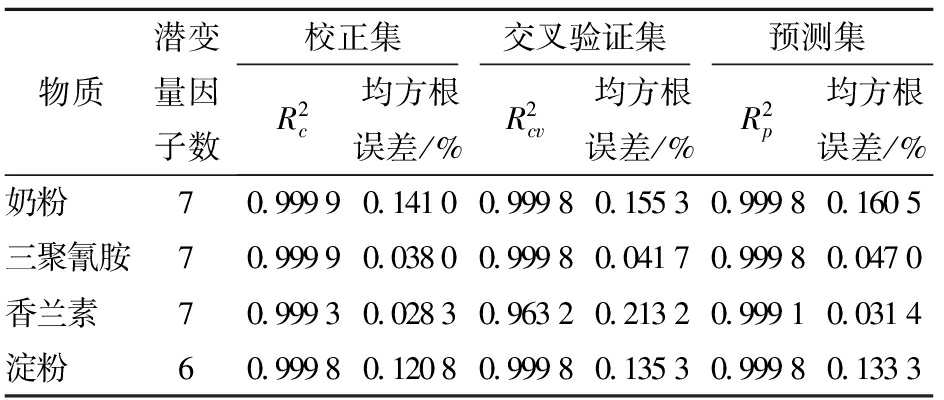
物质潜变量因子数校正集交叉验证集预测集R2c均方根误差/%R2cv均方根误差/%R2p均方根误差/%奶粉70.99990.14100.99980.15530.99980.1605三聚氰胺70.99990.03800.99980.04170.99980.0470香兰素70.99930.02830.96320.21320.99910.0314淀粉60.99980.12080.99980.13530.99980.1333
2.2.2 基于特征波长的PLS模型
利用两种特征波长筛选方法Laplacian score和ReliefF优选15个特征波长,并基于此设计多光谱PLS模型,结果如表4所示。所有模型决定系数均大于0.960 0,预测能力略低于全波长模型,但仍能较理想地预测奶粉、三聚氰胺、香兰素和淀粉。在奶粉、三聚氰胺和淀粉预测中,ReliefF-PLS模型效果更好,在香兰素预测中,Laplacian score-PLS模型效果更好。
表4 基于特征波长的多光谱PLS模型预测结果
Tab.4 Prediction results of multispectral PLS models based on characteristic wavelengths
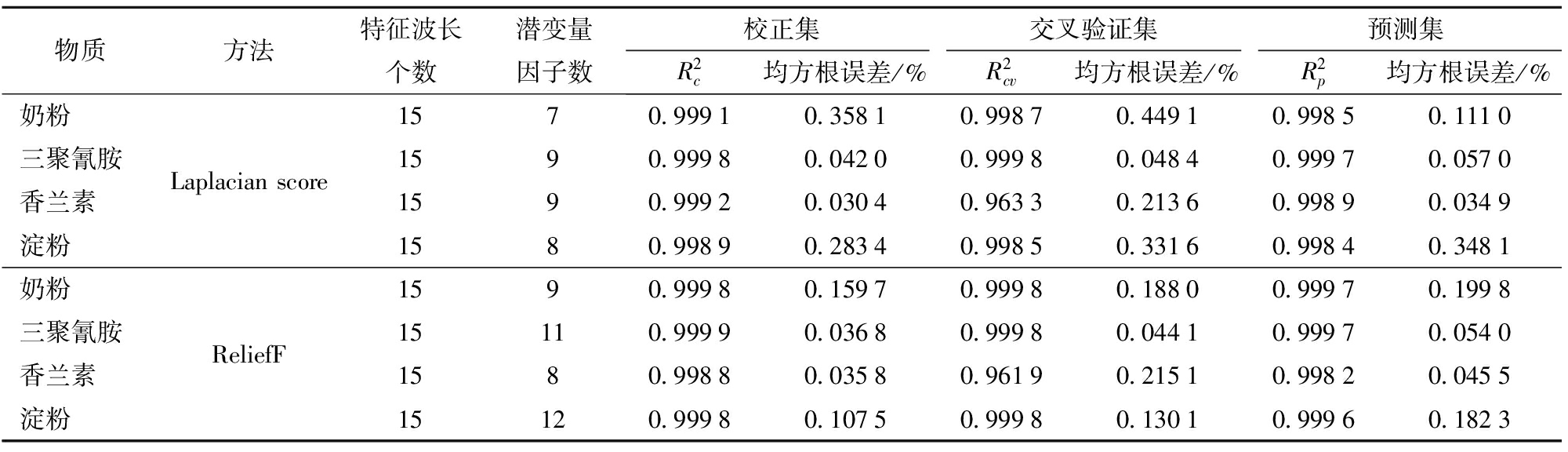
物质方法特征波长个数潜变量因子数校正集交叉验证集预测集R2c均方根误差/%R2cv均方根误差/%R2p均方根误差/%奶粉三聚氰胺香兰素淀粉Laplacianscore1570.99910.35810.99870.44910.99850.11101590.99980.04200.99980.04840.99970.05701590.99920.03040.96330.21360.99890.03491580.99890.28340.99850.33160.99840.3481奶粉三聚氰胺香兰素淀粉ReliefF1590.99980.15970.99980.18800.99970.199815110.99990.03680.99980.04410.99970.05401580.99880.03580.96190.21510.99820.045515120.99980.10750.99980.13010.99960.1823
与全波段PLS模型相比,采用筛选特征波长的方法会大大减小建模波长数量,进而缩减模型运算时间,提高检测效率。Laplacian score和ReliefF方法分别在奶粉、三聚氰胺、香兰素和淀粉检测模型中筛选出的波长在平均光谱上的分布如图3所示。由于Laplacian score为非监督算法,故对不同物质筛选出的波长基本相同,微小差异是由建立不同物质的检测模型时随机划分的不同训练集导致。所选最优波长点大多分布在1 450~1 550 nm之间特征吸收峰附近,以及1 680~1 700 nm之间。Laplacian score筛选了1 467、1 492、1 524 nm共3个与三聚氰胺最密切相关的吸收特征(见2.1节)。ReliefF筛选出的特征波长集中分布于1 213~1 241 nm、1 386~1 393 nm、1 428~1 456 nm以及 1 527~1 541 nm。其中,波长1 224 nm与香兰素中的C—H键有关[24],波长1 393、1 446、1 450 nm与淀粉中的O—H键的拉伸振动有关[25-27]。不同物质模型所对应的特征波长分布较接近。Laplacian score和ReliefF方法均挑选2.1节中讨论过的特征吸收峰 1 213 nm和 1 524 nm附近波长。
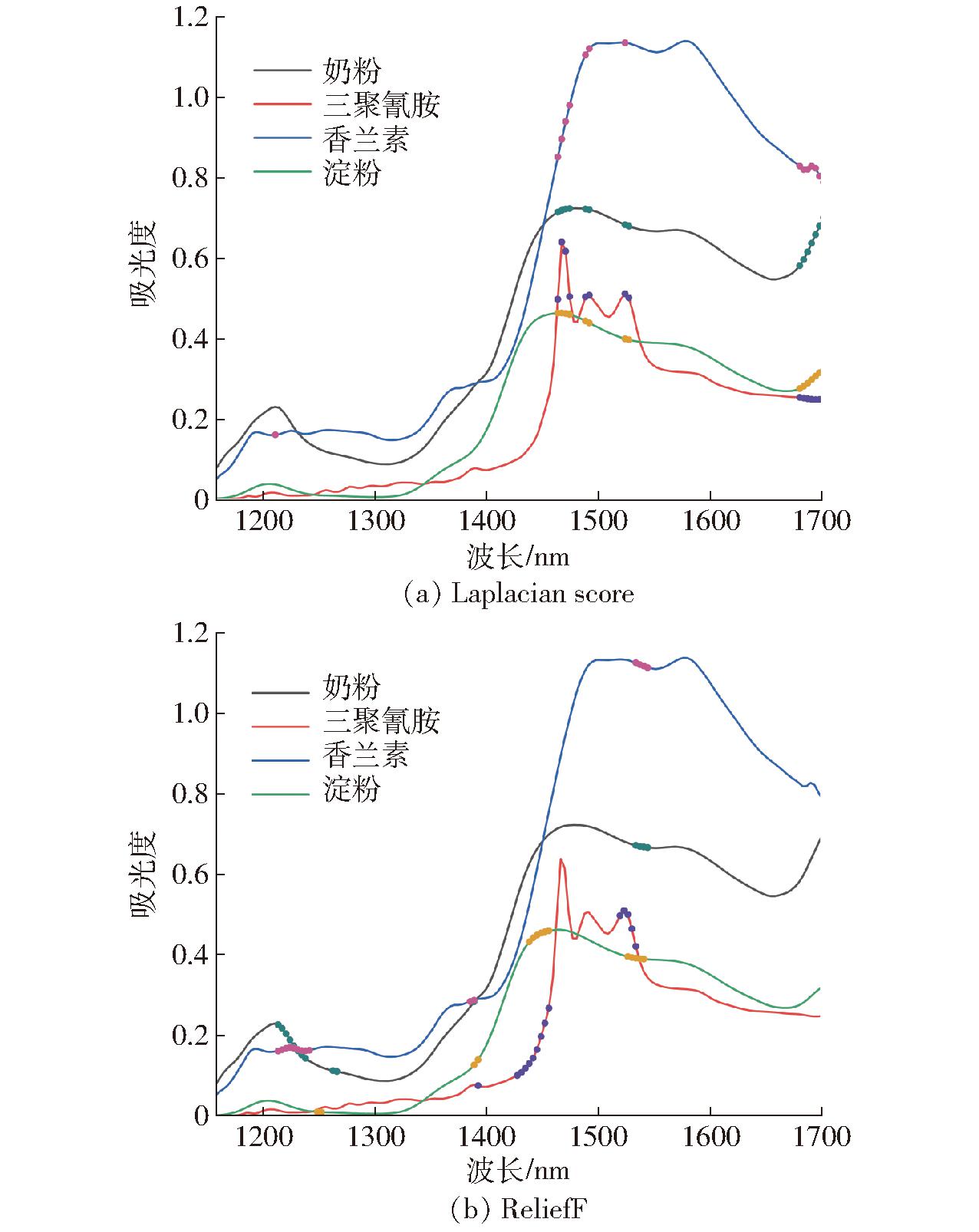
图3 特征波长在平均光谱图上的分布
Fig.3 Distributions of characteristic wavelengths on average spectra
将每种混合类型的27条光谱按照比例2∶1∶1随机划分为训练集、验证集和预测集,共得到训练集70条光谱(14条×5种),验证集35条光谱(7条×5种),以及预测集30条光谱(6条×5种)。奶粉、三聚氰胺、香兰素和淀粉基于全波段建立的CNN模型的预测结果如表5所示。所有模型决定系数均在0.995 0以上,预测效果略低于全波长PLS模型和多光谱PLS模型,具有较好的预测性能。根据自定义选择层的权重系数绝对值,优选前15个特征波长,并标注在平均光谱图上(图4)。
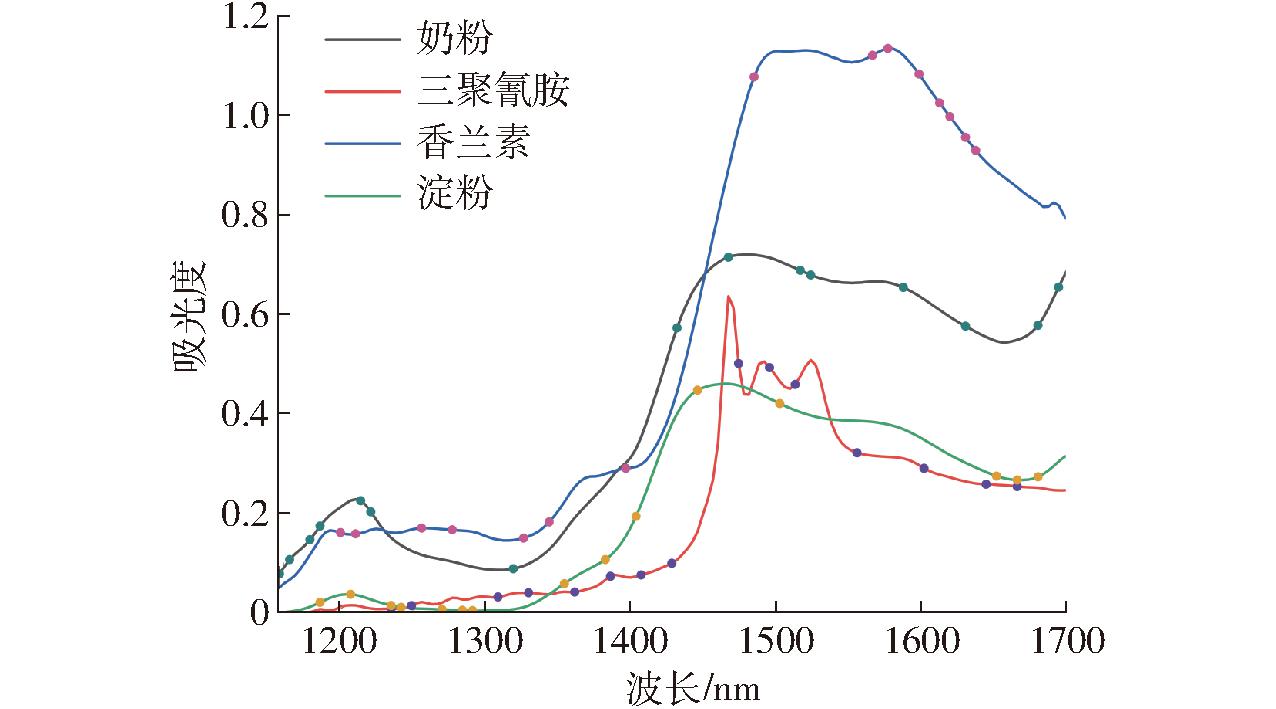
图4 CNN模型自定义层挑选的特征波长在平均光谱图上的分布
Fig.4 Distribution of characteristic wavelengths selected by self-defined layer in CNN model on average spectra
表5 CNN模型的预测结果
Tab.5 Prediction results of CNN model
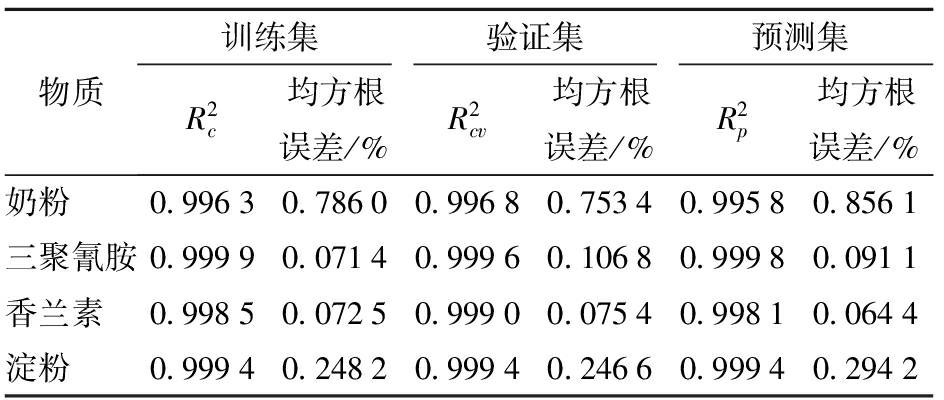
物质训练集验证集预测集R2c均方根误差/%R2cv均方根误差/%R2p均方根误差/%奶粉0.99630.78600.99680.75340.99580.8561三聚氰胺0.99990.07140.99960.10680.99980.0911香兰素0.99850.07250.99900.07540.99810.0644淀粉0.99940.24820.99940.24660.99940.2942
对比图3和图4可得CNN模型对应筛选出的15个波长点的分布较Laplacian score和ReliefF方法更分散。对于奶粉预测模型,Laplacian score、ReliefF和自定义层均选了1 524 nm作为特征变量。对于三聚氰胺预测模型,Laplacian score和自定义层均选了1 474 nm,文献[28]表明该波长与N—H键的第二泛音有关。对于香兰素预测模型,Laplacian score和自定义层均选了1 210 nm(C—H振动)[29]。对于淀粉预测模型,Laplacian score和自定义层均选了1 680 nm,ReliefF和自定义层均选了1 446 nm,文献[30]在马铃薯淀粉的掺假检测研究中同样筛选了1 446 nm为特征波长。
因ReliefF-PLS模型建模所需变量数少,且效果较优,故将包含空间信息像素点的光谱输入ReliefF-PLS模型,对其预测结果进行可视化。具体地,从SNV预处理后的子图像中随机选取150像素×150像素尺寸的ROI,提取ROI中每个像素点的光谱数据。以每个像素点作为一个样本点,构建尺寸为22 500×155的二维数据矩阵。提取ReliefF筛选的特征波长下数据输入构建好的相应多光谱PLS模型,得到每个像素点对应的物质预测质量分数一维向量结果(22 500×1)。将该向量重构为150×150矩阵数据,并制图,得到不同物质质量分数的可视化结果,如图5所示。图5中不同颜色变化表示模型对相应物质预测质量分数变化。图5a显示模型对质量分数62%、79%、85%和94.5%奶粉的可视化图由蓝到浅绿到黄绿变化明显,质量分数62%奶粉结果图中蓝色像素点多,表明预测质量分数主要集中在大约65%附近,质量分数94.5%奶粉结果图中主要为黄绿色,表明预测质量分数结果集中在大约95%附近。图5b显示掺有质量分数0、5%和10%三聚氰胺的样本的可视化图由黄到红变化明显,图5c显示掺有质量分数0、0.5%、1%和3%香兰素的样本的可视化图由浅蓝到绿到黄变化明显,图5d显示掺有质量分数0、5%、10%、15%和25%淀粉样本的可视化图由紫到红再到黄变化明显。以上表明模型具有良好的预测性能。
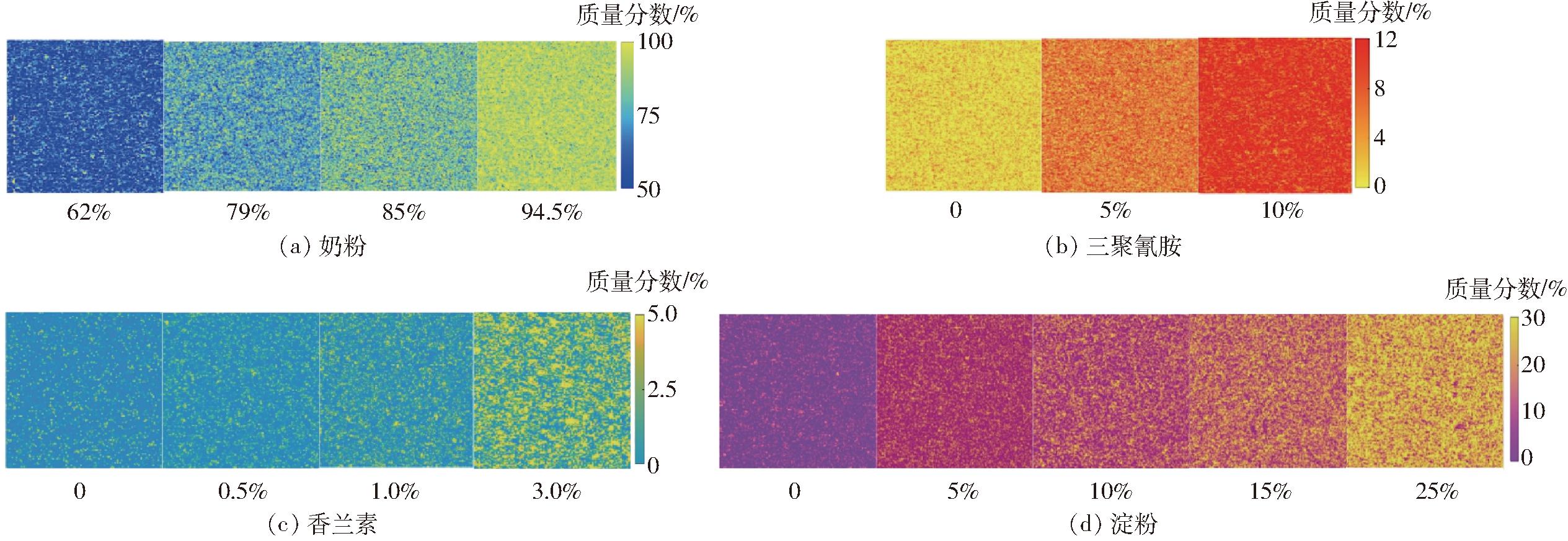
图5 基于ReliefF-PLS模型的4种物质含量预测的可视化图
Fig.5 Visualization of predicted contents of four substances based on ReliefF-PLS models
对采集的外部验证集和新外部验证集的高光谱图像数据应用相同的预处理,然后提取ROI平均光谱数据,获得外部验证集和新外部验证集各45条光谱(9条×5种)。提取ReliefF筛选的特征波长下的数据输入之前构建好的多光谱PLS模型。模型对预测集、外部验证集和新外部验证集的预测结果如表6所示。
表6 ReliefF-PLS多光谱模型对不同数据集的预测结果
Tab.6 Prediction results of ReliefF-PLS multispectral model for different datasets
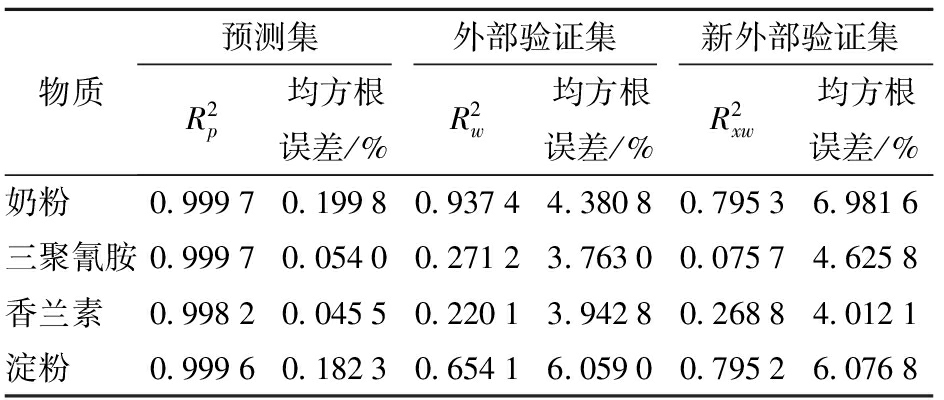
物质预测集外部验证集新外部验证集R2p均方根误差/%R2w均方根误差/%R2xw均方根误差/%奶粉0.99970.19980.93744.38080.79536.9816三聚氰胺0.99970.05400.27123.76300.07574.6258香兰素0.99820.04550.22013.94280.26884.0121淀粉0.99960.18230.65416.05900.79526.0768
尽管相比预测集,模型对外部验证集中的奶粉和淀粉的预测精度有所降低(R2由原来的0.999 7和0.999 6降为0.937 4和0.654 1;均方根误差由原来的0.199 8%和0.182 3%增长为4.380 8%和6.059 0%),但奶粉仍获得较理想的预测结果,淀粉的预测表现一般。精度降低可能是由外部验证集中奶粉、淀粉与建模样本集中奶粉、淀粉产品的批次等因素引起的样本差异导致的。模型对外部验证集中的三聚氰胺和香兰素预测结果差,R2只有0.2左右。原因可能是两者的掺假质量分数较低,批次等样品差异严重干扰了不同质量分数导致的光谱变化信息,从而影响了模型预测效果。
相比预测集,模型对新外部验证集中的奶粉和淀粉的预测精度降低,对三聚氰胺和香兰素的预测结果差。造成以上结果的原因除了不同批次因素外,还包括不同奶粉品牌之间的成分含量差异引起的光谱数据的干扰变动。然而,批次和品牌因素的综合影响是复杂的,体现在奶粉和三聚氰胺的![]() 明显小于
明显小于![]() 而香兰素的
而香兰素的![]() 与
与![]() 的结果相近,淀粉的
的结果相近,淀粉的![]() 却优于
却优于![]() 尽管如此,奶粉、三聚氰胺、香兰素和淀粉的新外部验证集均方根误差均大于外部验证集的均方根误差,这也说明不能单独依据R2评估模型的预测能力。模型对新外部验证集中奶粉和淀粉的预测结果表明,模型具有预测新品牌底物掺假样本的潜力,但预测精度仍需采用校正方法或其他措施进一步提高。
尽管如此,奶粉、三聚氰胺、香兰素和淀粉的新外部验证集均方根误差均大于外部验证集的均方根误差,这也说明不能单独依据R2评估模型的预测能力。模型对新外部验证集中奶粉和淀粉的预测结果表明,模型具有预测新品牌底物掺假样本的潜力,但预测精度仍需采用校正方法或其他措施进一步提高。
通过高光谱成像对婴幼儿奶粉中的多种掺假物(三聚氰胺、香兰素和淀粉)进行了定量检测分析。对预处理后的高光谱图像提取ROI及其平均光谱,然后在全波段建立PLS模型。采用Laplacian score(无监督)和ReliefF(有监督)两种特征波长筛选方法,并结合PLS算法建立多光谱预测模型。基于全波长数据训练包含自定义波长选择层的一维卷积神经网络模型。对Laplacian score、ReliefF和自定义选择层挑选的前15个重要波长进行对比分析。结果表明,与1DCNN模型相比,全波长PLS模型效果更佳,其预测集中奶粉、三聚氰胺、香兰素和淀粉质量分数预测结果的均方根误差分别为0.173 6%、0.047 0%、0.031 4%和0.133 3%;在基于特征波长的多光谱模型中,ReliefF-PLS表现出更好的预测效果,其预测集中奶粉、三聚氰胺、香兰素和淀粉质量分数预测结果的均方根误差分别为0.199 8%、0.054 0%、0.045 5%和0.182 3%。最后基于ReliefF-PLS模型预测结果进行可视化分析,同一物质不同浓度的可视化图颜色变化明显。采用两种外部验证集对模型进行了评估,模型对具有大质量分数的奶粉和淀粉预测结果较好。研究表明,高光谱成像技术可实现对于奶粉中同时掺入三聚氰胺、香兰素和淀粉3种掺假物含量的快速无损检测。
[1] NAGRAIK R,SHARMA A,KUMAR D,et al.Milk adulterant detection:conventional and biosensor based approaches:a review[J].Sensing and Bio-Sensing Research,2021,33:100433.
[2] 季美泉,费晓庆,丁涛,等.高效液相色谱法鉴别蜂蜜中掺入蜂王浆来源的蛋白质[J].食品安全质量检测学报,2020,11(1):153-157.
JI Meiquan,FEI Xiaoqing,DING Tao,et al.Identification of proteins from honey adulterated with royal jelly by high performance liquid chromatography[J].Journal of Food Safety &Quality,2020,11(1):153-157.(in Chinese)
[3] 孙超仁,王凤玲,王玉玮,等.基于特征脂肪酸变化对花生油掺伪快速鉴别方法研究[J].食品与发酵工业,2023,49(3):296-300.
SUN Chaoren,WANG Fengling,WANG Yuwei,et al.Rapid identification of adulteration in peanut oil based on the changes of characteristic fatty acid[J].Food and Fermentation Industries,2023,49(3):296-300.(in Chinese)
[4] 侯颖烨,王志元,谢建军,等.元素分析-稳定同位素质谱法结合化学计量学鉴别橄榄油掺假[J].中国油脂,2023,48(6):73-78.
HOU Yingye,WANG Zhiyuan,XIE Jianjun,et al.Olive oil adulteration identification using elemental analysis-stable isotope ratio mass spectrometer coupled with chemometrics[J].China Oils and Fats,2023,48(6):73-78.(in Chinese)
[5] TRIMBOLI F,COSTANZO N,LOPREIATO V,et al.Detection of buffalo milk adulteration with cow milk by capillary electrophoresis analysis[J].Journal of Dairy Science,2019,102(7):5962-5970.
[6] 胡珂青,刘欣悦,白金泽,等.基于同步荧光光谱的杜仲籽油掺假检测研究[J].中国油脂,2019,44(7):103-106.
HU Keqing,LIU Xinyue,BAI Jinze,et al.Adulteration detection of Eucommia ulmoides seed oil using synchronous fluorescence spectroscopy[J].China Oils and Fats,2019,44(7):103-106.(in Chinese)
[7] 赵茂程,吴泽本,汪希伟,等.基于光谱成像的猪肉新鲜度空间分布预测评价方法[J].农业机械学报,2022,53(3):412-422.
ZHAO Maocheng,WU Zeben,WANG Xiwei,et al.Evaluation of spectral imaging-based spatial predictions of freshness spatial distribution over pork[J].Transactions of the Chinese Society for Agricultural Machinery,2022,53(3):412-422.(in Chinese)
[8] 曹益飞,徐焕良,吴玉强,等.基于时序高光谱和多任务学习的水稻病害早期预测研究[J].农业机械学报,2022,53(11):288-298.
CAO Yifei,XU Huanliang,WU Yuqiang,et al.Early forecasting of rice diseases based on time series hyperspectral imaging and multi-task learning[J].Transactions of the Chinese Society for Agricultural Machinery,2022,53(11):288-298.(in Chinese)
[9] 王春雷,陈婉芝,卢彩云,等.基于高光谱成像的玉米收获后根茬行分割方法[J].农业机械学报,2020,51(增刊2):421-426.
WANG Chunlei,CHEN Wanzhi,LU Caiyun,et al.Segmentation method of maize stubble row based on hyperspectral imaging[J].Transactions of the Chinese Society for Agricultural Machinery,2020,51(Supp.2):421-426.(in Chinese)
[10] GUO L B,YU Y X,YU H Y,et al.Rapid quantitative analysis of adulterated rice with partial least squares regression using hyperspectral imaging system[J].Journal of the Science of Food and Agriculture,2019,99(12):5558-5564.
[11] 王伟,赵昕,褚璇,等.基于可见/近红外高光谱的八角茴香与莽草无损鉴别[J].农业机械学报,2019,50(11):373-379.
WANG Wei,ZHAO Xin,CHU Xuan,et al.Nondestructive identification of star anise and shikimmi by visible/near infrared hyperspectral images[J].Transactions of the Chinese Society for Agricultural Machinery,2019,50(11):373-379.(in Chinese)
[12] LIM J,KIM G,MO C,et al.Detection of melamine in milk powders using near-infrared hyperspectral imaging combined with regression coefficient of partial least square regression model[J].Talanta,2016,151:183-191.
[13] FORCHETTI D A P,POPPI R J.Use of NIR hyperspectral imaging and multivariate curve resolution (MCR) for detection and quantification of adulterants in milk powder[J].LWT—Food Science and Technology,2017,76:337-343.
[14] LIU Y S,ZHOU S B,HAN W,et al.Detection of adulteration in infant formula based on ensemble convolutional neural network and near-infrared spectroscopy[J].Foods,2021,10(4):785.
[15] ZHOU L,TAN L H,ZHANG C,et al.A portable NIR-system for mixture powdery food analysis using deep learning[J].LWT,2022,153:112456.
[16] ZHAO X,LI C H,ZHAO Z L,et al.Generic models for rapid detection of vanillin and melamine adulterated in infant formulas from diverse brands based on near-infrared hyperspectral imaging[J].Infrared Physics &Technology,2021,116:103745.
[17] LENG T,LI F,CHEN Y,et al.Fast quantification of total volatile basic nitrogen (TVB-N) content in beef and pork by near-infrared spectroscopy:comparison of SVR and PLS model[J].Meat Science,2021,180:108559.
[18] ROFFO G,MELZI S,CASTELLANI U,et al.Infinite latent feature selection:a probabilistic latent graph-based ranking approach[C]∥Proceedings of the IEEE International Conference on Computer Vision,2017:1398-1406.
[19] HE X F,CAI D,NIYOGI P.Laplacian score for feature selection[C]∥WEISS Y,SCHÖLKOPF B,PLATT J.Advances in Neural Information Processing Systems 18:Proceedings of the 2005 Conference.Nineteeth Annual Conference on Neural Information Processing Systems (NIPS 2005).Cambridge:MIT Press,2005:507-514.
[20] 项颂阳,许章华,张艺伟,等.高光谱图像分类的ReliefF-RFE特征选择算法构建与应用[J].光谱学与光谱分析,2022,42(10):3283-3290.
XIANG Songyang,XU Zhanghua,ZHANG Yiwei,et al.Construction and application of ReliefF-RFE feature selection algorithm for hyperspectral image classification[J].Spectroscopy and Spectral Analysis,2022,42(10):3283-3290.(in Chinese)
[21] KANG R X,WANG X,ZHAO M,et al.A comparison of benchtop and micro NIR spectrometers for infant milk formula powder storage time discrimination and particle size prediction using chemometrics and denoising methods[J].Journal of Food Engineering,2022,329:111087.
[22] FU X P,KIM M S,CHAO K L,et al.Detection of melamine in milk powders based on NIR hyperspectral imaging and spectral similarity analyses[J].Journal of Food Engineering,2014,124:97-104.
[23] MAUSER L J,CHERNYSHOVA A A,HIATT A,et al.Melamine detection in infant formula powder using near-and mid-infrared spectroscopy[J].Journal of Agricultural and Food Chemistry,2009,57(10):3974-3980.
[24] HOFFMAN L,INGLE P,KHOLE A H,et al.Discrimination of lamb (Ovis aries),emu (Dromaius novaehollandiae),camel (Camelus dromedarius) and beef (Bos taurus) binary mixtures using a portable near infrared instrument combined with chemometrics[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy,2023,294:122506.
[25] BABAEE B,KHANMOHAMMADI M,GARMARUDI A B,et al.Effect of peeling and point of spectral recording on sucrose determination in sugar beet root using near infrared spectroscopy[J].Infrared Physics &Technology,2019,103:103065.
[26] DELANEY J K,CONOVER D M,DOOLEY K A,et al.Integrated X-ray fluorescence and diffuse visible-to-near-infrared reflectance scanner for standoff elemental and molecular spectroscopic imaging of paints and works on paper[J].Heritage Science,2018,6:31.
[27] ZHAO Y R,YU K Q,LI X,et al.Detection of fungus infection on petals of rapeseed (Brassica napus L.) using NIR hyperspectral imaging[J].Scientific Reports,2016,6(1):38878.
[28] LABORDE A,PUIG-CASTELLV F,BOUVERESSE D J R,et al.Detection of chocolate powder adulteration with peanut using near-infrared hyperspectral imaging and multivariate curve resolution[J].Food Control,2021,119:107454.
F,BOUVERESSE D J R,et al.Detection of chocolate powder adulteration with peanut using near-infrared hyperspectral imaging and multivariate curve resolution[J].Food Control,2021,119:107454.
[29] SANTOS I A,CONCEIÇ O D G,VIANA M B,et al.NIR and MIR spectroscopy for quick detection of the adulteration of cocoa content in chocolates[J].Food Chemistry,2021,349:129095.
O D G,VIANA M B,et al.NIR and MIR spectroscopy for quick detection of the adulteration of cocoa content in chocolates[J].Food Chemistry,2021,349:129095.
[30] ZHAO X,WANG W,CHU X,et al.Rapid and nondestructive quantification of cassava starch adulterants in potato starch by using hyperspectral imaging[C]∥2018 ASABE Annual International Meeting.American Society of Agricultural and Biological Engineers,2018.